Introduction
한우는 5,000년 역사를 지닌 한국 고유의 품종으로 열악한 환경에 잘 적응하며, 특히 근내지방과 같은 육질이 우수하다고 알려져 있는 중요한 유전자원이다(Hwang et al., 2010). 과거 우리나라가 농경사회였을 때는 역우(draft animal)로 주로 사용되었으나, 한국사회가 빠르게 발전하면서 농업 기계화가 빠르게 진행되는 과정에 한우의 역할이 역우에서 고기소로 바뀌게 되었다(Lee et al., 2014). 이에 따라, 1963년 한우개량협의회의 결정에 따라 한우개량에 대한 논의가 진행되고, 이에 따라 역우에서 육용우로의 전환기를 맞이하였으며, 육용종으로서 한우를 개량하기 위하여 처음으로 개량 목표를 설정하고 현재까지 한우개량을 진행해 오고 있다(Park et al., 2013; Lee et al., 2014).
한우개량 목표는 농림축산식품부 축산법 MAFRA (2017)에서 정하는 12개월 체중, 도체중(carcass weight), 근내지방도(marbling score), 등지방두께(back fat thickness) 그리고 등심단면적(eyemuscle area)과 같은 경제적으로 유용한 도체형질을 중심으로 농림축산식품부 축산법에 고시하고 있다(Park et al., 2013; Kim et al., 2017). 한우개량목표에 따라 한우개량계획은 당대 및 후대검정방법을 이용하여 목표형질의 추정육종가를 산출한다(Park et al., 2013; Kim et al., 2017). 당, 후대 검정방법을 통한 육종가의 추정은 혈통정보, 당, 후대검정에서 측정한 도체형질에 대한 표현형, 그리고 최근에는 유전체 정보(genomic information; 50 K chip)를 개체모형(animal model; best linear unbiased prediction [BLUP])에 적용하여 아비, 어미의 능력 혹은 아직 태어나지 않은 자손의 능력을 미리 예측할 수 있다(Hayes and Goddard, 2001; Lee et al., 2014; Chung et al., 2018). 한우의 검정방법은 국가에서 주도하는 우량 씨수소와 씨암소 사이에서 태어난 수송아지의 능력을 12개월 체중을 중심으로 검정하는 당대검정(performance test)과 당대검정에서 선발된 후보 씨수소의 후대의 성적을 검정하여 보증 씨수소를 선발하는 후대검정(progeny test)이 있다. 또한, Yun et al. (2016)의 연구에서는 한우의 후대 정보를 이용하여서 성장능력에 대한 성장곡선을 추정하기도 하였다. 현행 한우개량은 이들 두 검정방법을 통하여 우수한 보증 씨수소를 선발하여 농가에 보급하고 있다(Park et al., 2013; Kim et al., 2017; Chung et al., 2018).
이러한 당, 후대 검정(performance and progeny test)방법은 우량 가축을 선발하는데 매우 효율적인 방법이지만 시간과 비용이 많이 소요되는 단점도 가지고 있다(Lee et al., 2013; Park et al., 2013; Lee et al., 2014). 더욱이 시간 및 비용뿐 만 아니라, 농가에 보급되는 과정에서 추정 육종가의 정확도는 매우 중요한 요소일 것이다. 따라서, 본 연구의 목적은, 후대검정이라는 방법을 이용한 선발육종에서 검정에 참여하는 후대의 수 및 아비(sire)의 수에 따라서 추정육종가의 정확도가 어떻게 변하고 있으며, 아울러 후대검정에 참여하는 개체의 혈통정보의 수(pedigree depth)가 추정육종가의 정확도에 미치는 영향을 조사하기 위하여 수행 되었다.
Materials and Methods
실험 설계
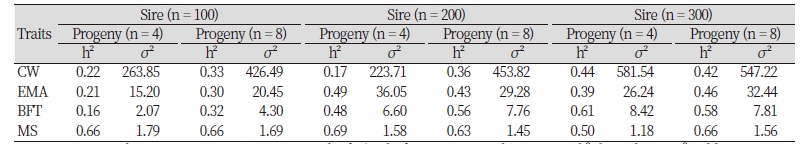
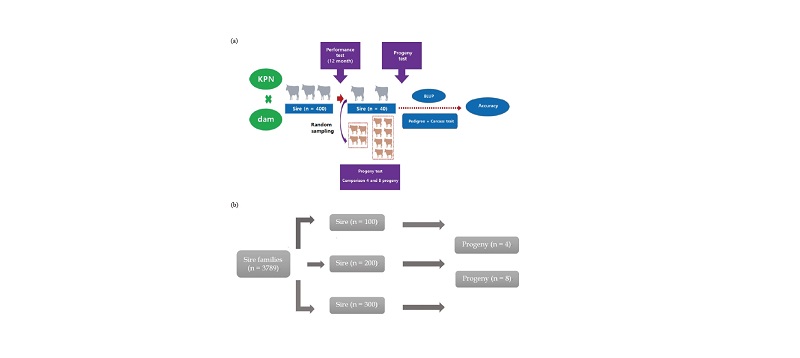
본 연구의 목적인 선발육종에서 검정에 참여하는 후대의 수와 혈통정보의 수(pedigree depth)가 후보 씨수소 추정육종가의 정확도에 미치는 영향을 분석하기 위해서 국가단위 한우 개량사업(당, 후대검정)에서 제공한56,383두의 혈통정보와 16,740두에 대한 도체형질 표현형, 즉 도체중(carcass weight; CW), 배최장근단면적(eye muscle area; EMA), 등지방두께(back fat thickness; BFT) 그리고 근내지방도(marbling score; MS9) 등 4개의 도체형질에 대한 표현형 정보를 활용하였다. Fig. 1에서 보여주는 것과 같이, 사용된 표본(sample)은 도체형질 표현형 자료가 모두 있는 씨수소 혹은 후보씨수소로 사용된 전체 3,789 두를 추출한 후, 이들의 후대의 자손수가 추정 육종가(estimated breeding value; EBV) 정확도에 미치는 영향을 알아보기 위하여 8 마리 이상의 자손을 가지는 씨수소를 각각 100두, 200두 그리고 300두씩 임의 추출(random sampling)한 후에 각각의 sire 개체에서 자손을 4마리, 8마리씩 추출하였다. 같은 아비를 가지는 자손들은 서로 반형매(half-sibs) 관계에 있으며 보다 정확한 비교를 위해서 같은 두 수의 씨수소 그룹에서 임의 추출한 4마리의 자손을 8마리 추출 시 포함시켜 주었다. 따라서, 전체 분석에 사용된 자손의 수는 sire (n) × progeny (n) 마리로, 순서대로 400, 800, 800, 1600, 1200, 2400마리의 progeny 데이터를 이용해서 분석을 진행하였다. 또한 혈통의 수(pedigree depth)가 추정육종가의 정확도에 영향을 미치는 요인을 살펴보기 위하여, 10개의 반형매(half-sib) 가계를 무작위 추출하였고, 이들 10개의 반형매 집단의 총 혈통(12세대)을 이용하였을 때의 정확도와, 동일 10개의 반형매 집단의 혈통 1세대만을 이용하였을 때의 정확도를 비교 분석하였다.
공시재료
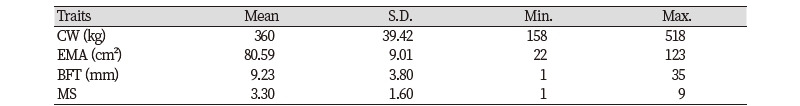
본 연구에서 사용된 16,740두에 대한 도체중, 등심단면적, 등지방두께 및 근내지방도의 기술 통계량 값을 Table 1에 나타내었다. 각 형질의 평균 및 표준편차는 각각 616.5 ± 63.76 kg, 360 ± 39.42 kg, 80.59 ± 9.01 cm², 9.23 ± 3.80 mm, 3.3 ± 1.6점으로 추정되었고, Kim et al. (2010)이 734두의 후대 검정우 기술 통계량값으로 보고한 356.5 ± 38.2 kg, 77.7 ± 8.2 cm2, 10.3 ± 4.0 mm 및 3.3 ± 1.7(순서대로 도체중, 배최장근 단면적, 등지방두께, 근내지방도)의 값과 비슷한 경향을 보여주었다.
육종가 추정
각 형질에 대한 개체의 추정 육종가는 다음의 혼합선형모형을 이용하여 산출하였다.
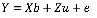 (1)
(1)
Y는 개체 별 표현형 관측치를 나타내는 벡터X는 출생년도(2003 - 2013) 및 월(1, 4, 5, 6, 7, 10, 11, 12), 그리고 차수(36 - 56) 등에 따른 표현형 보정을 위한 고정 효과 벡터이며 Z는 혈통 정보를 기반으로 개체간 관계를 설명하는 임의 효과 벡터이며 b와 u는 각각 고정 효과 추정치와 개체에 대한 추정치 벡터이고 e는 임의의 오차이다. 육종가 추정을 위하여 혈통정보를 연대기순으로 정리하기 위하여 pedigree viewer (The University of New England, New England, Australia)를 사용하였고 (Kinghorn and Kinghorn, 2010), 육종가 추정은 ASReml (4.1 version, VSN International, Hermel Hempstead, United Kingdom)을 활용하였다 (Gilmour et al., 2015).
Results and Discussion
유전력(heritability) 추정
유전분산(genetic variance; Vg), 잔차분산(residual variance; Ve) 및 표현형분산(phenotypic variance; Vp)과 같은 분선성분을 이용하여서 추정한 유전력(heritability)과 유전분산에 대한 결과는 Table 2에 나타내었다. 유전력은 개체모형을 이용하여 추정한 육종가의 표준오차(PEV)를 이용한 이론적인 정확도를 추정하는데 영향을 미치는 중요한 요인으로 알려져 있다(Choi et al., 2017). 따라서 검정에 참여하는 후대의 수에 따라서 유전력의 변화를 추적하는 것은 매우 중요한 사항일 것이다. 본 연구에서 분석한 결과 검정에 참여하는 자손의 수가 많은 경우의 형질에 대한 유전력 추정치가 자손의 수가 적은 경우보다 높은 값을 보여주었다. 지방 형질로 표현되는 등지방두께(BFT)와 근내지방도(MS)의 유전력 평균값이 나머지 세개의 형질에서 보다 높은 값을 보여주었다(Table 2). 각 형질에 대한 유전력 추정의 결과 도체중에서는 0.32, 등심단면적은 0.38 그리고 등지방두께와 근내지방도에서는 각각 0.45, 0.63으로 추정되었다. 이러한 결과는 Choi et al. (2006)이 24개월 도축 자료를 분석한 유전력 추정치 결과값인 도체중(0.32), 등심단면적(0.33), 등지방두께(0.51), 근내지방도(0.5)의 값과 비교해 보았을 때, 등지방두께의 유전력 추정치는 다소 낮게, 등심단면적과 근내지방의 유전력 추정치는 비교적 높게 추정되었다. 등심단면적의 유전력 추정치가 차이가 나는 이유는 형질의 특성상, 등심을 절단 할 시에 발생하는 오차가 다른 형질의 오차보다 크고, 근내지방의 유전력 추정치에 차이가 나는 이유는 한우의 육종 계획을 수립할 때, 근내지방함량 증가에 초점을 맞춰 선발과 교배를 시행하기 때문이라고 사료된다.
추정육종가(EBV) 의 정확도 분석
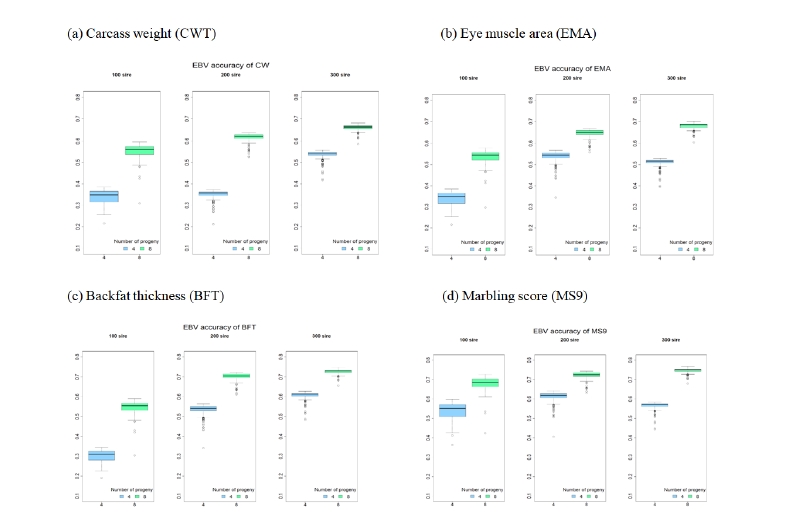
검정에 참여하는 후대의 수(n = 4, 8)에 따라서 변화하는 추정육종가의 정확도에 관한 결과를 Fig. 2와 Table 3에 제시하였다. 이번 분석에 사용된 한우들의 혈통자료와 각 개체에 대한 표현형자료를 통합하여서 BLUP으로 개체에 대한 유전능력평가를 수행해주었다. 각 형질에 대해서 자손 수와 씨수소 수에 따른 추정육종가 정확도 값의 변화를 Fig. 2에 나타내 주었다. 모든 형질에서 같은 수의 씨수소 일 때, 각 개체당 자손의 수가 4마리에서 8마리로 증가함에 따라 추정육종가 정확도 또한 확연하게 증가하는 것을 확인 할 수 있다. 더 정확한 비교를 위하여 Table 3에 모든 그룹에 대한 평균 추정육종가 정확도 값을 수치화 시켜서 제시하였다. 후대의 수가 4마리인 경우 정확도는 약 30 - 60%사이로 나타났고, 후대의 수가 8마리인 경우에는 약 50 - 75% 사이로 나타났다. 이는 최근 후대의 기록이 암소의 추정 육종가 정확도에 미치는 영향을 분석한 결과 후대의 수가 2 - 3마리일 때 약 40%, 후대의 수가 4마리인 경우 48 - 57%, 후대의 수가 5 - 6마리인 경우 50 - 60%의 결과값과 비슷하게 후대의 수가 증가 할수록 추정육종가의 정확도 또한 증가하는 경향을 보여주었다(Kim et al., 2010; Park et al., 2017).
|
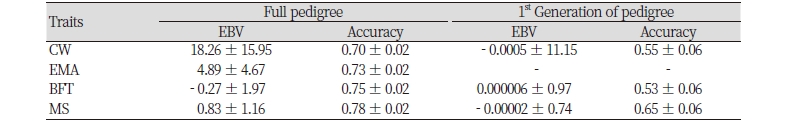
Table 3. Accuracy of estimated breeding value (EBV) on their number of progeny. 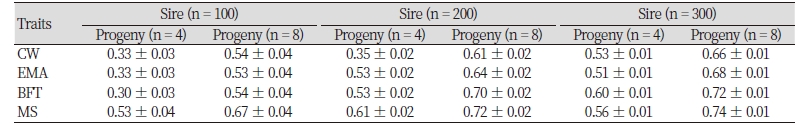
|
|
|
CW, carcass weight; EMA, eye muscle area; BFT, back fat thickness; MS, marbling score. |
|
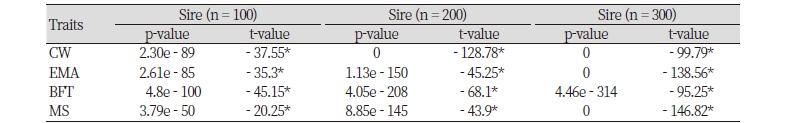
후대의 수에 따른 추정육종가 정확도 값이 유의적인 차이를 가지는지 확인하기 위해서 그룹간 등분산성 검정을 실시한 후에 독립표본 t검정을 시행해주었다. 검정결과에 대한 값은 Table 4에 제시하였다. Table 4에 나타낸 것과 같이 모든 형질에 대해서 p-value가 상당히 낮고, │t│값이 큰 결과를 보이는 것에 따라 그룹간 (progeny = 4, progeny = 8) 평균이 유의적으로 큰 차이가 난다는 것을 확인할 수 있다. p-value 값이 0으로 나온 결과는 실제 p-value가 상당히 낮은 값을 가짐으로 인해 발생한 결과이다. 이는 통계적으로 매우 유의미하다는 것을 의미하고, 실제로 그 값은 0을 나타내지 않는다.
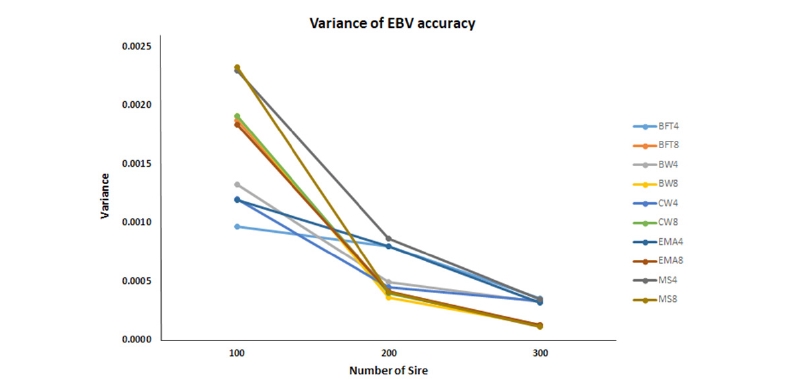
Fig. 3에는 모든 형질에 대한 추정육종가 정확도를 계산하기 위하여 분산성분 (variance component) 값을 씨수소의 수에 따라서 결과를 제시하였다. 보는 바와 같이, 개체의 수가 증가할수록 각 그룹에서 추정육종가 정확도의 분산 값이 감소하는 것을 확인 할 수 있다. 이 결과를 통해서 자손의 수뿐만이 아닌 분석을 하기 위한 씨수소 개체의 수 또한 정확도에 영향을 끼친다는 것을 알 수 있다. 따라서, 씨수소의 육종가를 추정할 경우에 추정육종가의 정확도를 높이기 위해서는 후대의 정보를 가능한 많이 확보하고 씨수소의 정보 또한 많이 확보하여 유전평가를 하는 것이 효과적인 방법일 것이다. 따라서, 지속적이고 정확한 표현형 및 혈통의 기록과 관리가 앞으로도 계속 되어야 하고 추정육종가 정확도의 상승효과를 위하여 계속해서 참조 집단의 크기를 늘리는 것이 중요한 부분이라고 여겨진다.
혈통정보의 수(pedigree depth)에 따른 추정육종가의 정확도 분석
혈통의 수(pedigree depth)에 따라서 추정 육종가(EBV)와 정확도가 어떻게 변화하는지를 살펴보기 위하여 무작위 추출된 10개의 반형매(half-sib) 가계의 12세대 혈통 혹은 1세대 혈통을 이용하여 결과를 비교 하였다. 그 결과, 10개의 반형매 집단의 후대 8두와 12세대 혈통을 이용하여 추정된 육종가의 평균은 BFT에서 - 0.27, CW에서 18.26, EMA에서 4.89 그리고 MS에서 0.83이였고, 정확도는 0.75, 0.70, 0.73, 0.78 (순서대로 BFT, CW, EMA, MS)의 결과를 보여주었다. 반면, 동일 10개의 반형매집단의 후대 8두와 1세대 혈통만을 이용하여 추정된 육종가는 BFT에서 0.000006, CW에서 - 0.0005 그리고 MS에서 - 0.00002이였고, 정확도는 0.53, 0.55, 0.65 (순서대로 BFT, CW, MS)의 결과를 보여주었다. 추정육종가와 정확도에 대한 자세한 결과는 Table 5에 나타내었다. 다만, 1 세대(후대의 성적) 만을 이용하여 추정하는 과정에서 EMA 형질에 대하여 singularity가 발생하여서 해당 형질을 제외하고 분석을 진행하였다. 이러한 결과는 혈통정보의 품질(quality)과 수(numbers of pedigree)가 매우 큰 영향을 미치는 것으로 판단된다. 최근 국내 각 시도 농업기술센터 및 브랜드 한우를 중심으로 자체 보증씨수소를 선발하고자 하는 논의가 진행되고 있다. 즉 국가단위 한우개량사업을 중심으로 각 지자체 혹은 브랜드한우의 자체 후보 씨수소를 이용하여 씨수소를 선발하고자 할 때, 고려해야 하는 사항 중에서 혈통의 오류가 없는 혈통정보의 품질 및 혈통의 세대수가 매우 중요할 것이다. 농가 및 지자체에서는 혈통관리가 체계적이지 않아 정확하지 않은 혈통정보를 이용하여 개체의 능력을 추정하면 정확도는 물론이고 육종가에서도 매우 다른 결과가 나와 개체를 선발하는데 문제점이 될 수 있다. 이러한 문제점을 해결하기 위해서 최근 유전체정보를 기반으로한 유전체혈연관계(genomic relationship matrix; GRM)를 이용한다면 보다 정확한 혈통추적 및 육종가 추정이 가능할 것으로 사료된다.
Conclusion
본 연구는 국가단위 한우 개량사업(농협 한우개량사업소 및 축산물품질평가원)에서 제공받은 56,383두의 혈통정보와 16,740두의 표현형 정보를 이용하여서 씨수소의 유전능력을 추정하고 자손의 수가 추정육종가 정확도에 미치는 영향을 알아보고자 수행하였다. 유전능력 평과 결과, 자손의 수가 4마리인 경우 추정육종가의 정확도는 약 30 - 60%, 자손의 수가 8마리인 경우에는 약 50 - 75% 사이로 나타났다. 다시 말해서 자손의 수가 4마리에서 8마리로 증가 할 시, 추정육종가의 정확도 역시 15 - 20% 정도 증가하는 것을 보여주었다. 이와 별개의 결과로, 씨수소 수의 증가로 각 형질에 대한 추정육종가 정확도 관찰 값들 사이의 편차가 줄어들어 보다 일관된 육종가 추정이 가능하다는 것을 확인하였다. 결과적으로 씨수소의 추정육종가의 정확도를 올리기 위해서는 씨수소 자신의 직접정보뿐만이 아닌 각 씨수소 후대의 정보 및 수 또한 중요하다는 것을 확인하였다. 따라서, 후대 정보를 가능한 많이 확보하고 신중하게 관리하여 유전능력평가를 수행하는 것이 보다 정확한 결과를 가져다 줄 것으로 판단된다.
Acknowledgements
본 연구는 충남대학교 농업과학연구소 자체연구과제 연구비(농가한우암소개량을 위한 유전평가기법 개발)로 수행하였습니다. 연구비 지원에 감사 드립니다. 연구자료를 제공해 주신 국립축산과학원에도 감사 드립니다.
Authors Information
Sungbong Jang, Chungnam National University, Master student
So Yeon Kim, Chungnam National University, Master student
Soo-Hyun Lee, Chungnam National University, Ph.D. student
Min Gwan Shin, Chungnam National University, Researcher
Jimin Kang, Chungnam National University, Ph.D. student
Dooho Lee, Chungnam National University, Ph.D. student
Sidong Kim, National Institute of animal science, PhD
Seung Hee Noh, NongHyup, PhD
Tae Jeong Choi, National Institute of animal science, PhD
Seung Hwan Lee, https://orcid.org/0000-0003-1508-4887



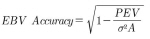 (2)
(2)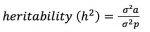 (3)
(3)