Introduction
대장균에서 최초로 보고된 clustered short repeat은 많은 미생물에서 공통적으로 존재한다는 것이 후속 연구를 통하여 증명되었으며, 이 서열을 clustered regularly interspaced short palindromic repeats (CRISPR) 로 명명하였다(Ishino et al., 1987; Jansen et al., 2002). 이후 Cas9이 CRISPR과 함께 작동하여 세균의 획득면역(adaptive immunity) 기구를 구성한다는 것을 알게 되었다(Barrangou et al., 2007). CRISPR/Cas9 시스템을 활용하여 연구자가 원하는 염기서열의 DNA를 이중가닥 절단 (double strand break, DSB) 할 수 있다는 것을 증명함으로써 유전자 편집 기술이 개발되었다(Jinek et al., 2012). Cong et al. (2013)은 이를 확대하여 미생물 뿐만 아니라 포유동물 세포에서도 CRISPR/Cas9에 의한 이중가닥 절단이 가능하며 비상동말단연결(nonhomologous end-joining, NHEJ)에 의한 수선과정 중에 발생하는 오류로 인해 특정 유전자의 원하는 위치에 결실 및 삽입 등을 유도할 수 있고, 상동재조합 (homology directed repair, HDR)에 의한 유전자 치환이 가능함을 증명하였다. 이후 여러 연구그룹이 다양한 작물에서 CRISPR/Cas9을 활용한 유전자 교정이 가능함을 보고하여 작물 분야에서 본격적인 유전자 교정시대가 열리게 되었다(Belhaj et al., 2015; Xie et al., 2015; Jiang et al., 2017; Miao et al., 2018).
CRISPR/Cas9에 의한 유전자 교정은 게놈내의 특정 DNA 염기서열을 인식하고 이중가닥절단을 유도하여 세포 내 비상동말단연결 수선기구의 오류에 의한 특정위치 염기서열의 삽입(insertion), 결실(deletion), 전좌(translocation)를 유도하여 특정 유전자의 기능을 상실하게 하는 것이 주요한 특징이다(Fig. 1). 특정 염기서열들을 목표 염기서열로 교체하는 엄밀한 의미의 유전자교정은 외래 DNA (donor)와 이중가닥 절단 위치의 상동재조합에 의한 수선기구에 의해서 발생한다. 상동재조합에 의한 수선 효율은 매우 낮으며 대부분의 유전자교정 위치에 삽입이나 결실이 발생할 가능성이 높아 보편적으로 널리 활용되고 있지는 않다. 특정 염기서열을 교정하기 위한 도구를 개발하기 위하여 연구자들은 CRISPR/Cas9가 게놈상의 특정 염기서열에 결합하는 특성과 다양한 추가적인 기술을 융합하여 연구자 의도대로 특정 염기를 변화시킬 수 있는 염기교정기술을 개발하여 단순한 유전자 돌연변이 유도가 아닌 정교한 유전자 염기 교정이 가능하게 되었다. 본 총설에서는 염기교정 기술 별 연구현황 및 작물에 적용현황에 대하여 고찰하고자 한다.
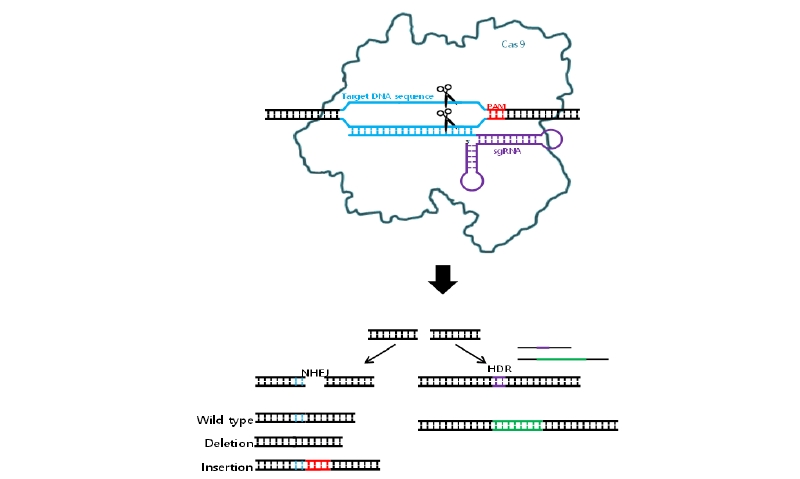
Fig. 1 Diagrams of gene editing mechanisms using CRISPR/Cas9. CRISPR/Cas9 cleave target sequence complementary to guide RNA. Cleaved DNA flagments are repaired through NHEJ or HDR mechanisms in living cells. The mis-repair during NHEJ process can cause deletion or addition mutations in target sequence. CRISPR, clustered regularly interspaced short palindromic repeats; NHEJ, nonhomologous end-joining; HDR, homology directed repair; PAM, protospacer adjacent motif; sgRNA, single guide RNA.
Cas9 핵산분해 효소 활성 돌연변이 개발
일반적인 유전자 교정에서 사용하는 Cas9은 DNA 이중 가닥을 잘라서 NHEJ 혹은 HDR을 유도하여 유전자의 기능을 상실하게 하는 능력을 가지고 있다. 그러나, 염기교정을 위해서는 guide RNA에 의해서 특정 염기서열을 인식하는 Cas9의 특성은 유지하면서 Cas9에 의한 이중가닥 절단 능력은 상실하도록 돌연변이를 제작하여야 한다. 이를 위해서 Cas9의 핵산 분해 활성에서 중요한 역할을 수행하는 RuvC1과 HNH 도메인에 D10A와 H840A 돌연변이를 제작하였다(Fig. 2A). 두 개의 돌연변이를 동시에 갖고 있는 Cas9은 핵산 분해 활성을 잃은 dead Cas9 (dCas9)이 되고, 각각의 돌연변이를 한 개씩만 갖고 있으면 DNA의 서로 다른 한 가닥을 자르는 두 가지 종류의 nick Cas9 (nCas9)이 된다(Jinek et al., 2012). 또한, 연구자들은 사용 목적에 따라 이들 돌연변이 Cas9에 다양한 종류의 effector를 결합하여 새로운 종류의 분자생물학적 도구를 개발하였다(Fig. 2B).
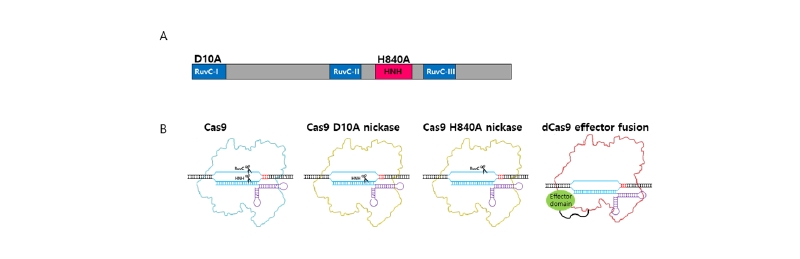
Fig. 2 Schematic representations of different kinds of Cas9 nuclease mutants. (A) Diagram of dCas9 or nCas9 developed by mutagenesis of Cas9 nuclease domains. 10th amino acid, D is changed to A in RuvC-I domain and 840th amino acid, H is changed to A in HNH domain. (B) Diagram of functional mechanisms of dCas9 and nCas9 mutants. Cas9 D10A make a nick to the strand to have complementary sequence to gRNA. Cas9 H840A make a nick to the strand to have sequence same to gRNA.
Cytosine base editor (CBE)의 개발 및 식물용 CBE의 개발
최초의 염기교정기술 개발은 cytosine을 thymine으로 치환시키는 cytosine base editor의 개발로부터 시작하였다(Komor et al., 2016). Cytosine base editor가 cytosine의 아민기를 제거하여 uracil로 만들면 일련의 세포 내 수선 기작에 의하여 uracil은 adenine으로 변환된다(Fig. 3). 현재까지 알려진 cytosine deaminase는 일반적으로 RNA에서 작동을 하지만 일부는 single strand DNA (ssDNA)에서도 작동할 수 있는 것으로 알려져 있다(Harris et al., 2002). Cas9이 DNA에 결합하면 이중나선을 풀어서 R-loop를 형성하고, guide RNA가 결합하지 않은 가닥의 ssDNA가 노출되므로 cytosine deaminase가 작동할 수 있게 된다. Komor et al. (2016)은 인간 activation-induced cytidine deaminase (AID), 인간 APOBEC3G, 쥐 APOBEC1 그리고 칠성장어 CDA1 4가지 종류의 cytosine deaminase를 이용하여 ssDNA 에 대한 탈아민 활성을 측정하였다. 가장 높은 활성을 보인 쥐 APOBEC1을 dCas9의 아미노 말단에 결합시켜 탈아민 활성이 있는지 확인하였다. 또한, dCas9과 APOBEC1을 연결시키는 다양한 링커를 검토한 결과, XTEN 링커(16 아미노산 잔기)가 탈아민 활성 및 탈아민 염기범위에서 최적의 효율을 나타냈다(Komor et al., 2016). 이러한 과정을 거쳐 APOBEC1-XTEN-dCas9으로 구성된 최초의 base editor가 개발되었으며, 이를 BE1이라고 명명하였다 (Komor et al., 2016). BE1의 염기교정 효율은 인간 세포주에서는 0.8 - 7.7%로 매우 낮은 효율을 보였으나 시험관내에서는 효율이 5 - 36 배까지 증가하였다. 세포 내에는 uracil DNA glycosylase (UDG)가 세포내의 uracil DNA 염기를 제거하고 이를 수선하는 base-excision repair (BER) 시스템이 존재하여 효율이 낮은 것으로 생각되어, 박테리오파지 PBS1에서 유래한 83개 아미노산으로 구성된 uracil DNA glycosylase inhibitor (UGI)를 BE1의 카르복실 말단에 결합하여 BE2를 개발하였다. BE2는 인간 세포주인 HEK293 T세포에서 cytosine base editing 효율이 20%에 이르게 되었다(Komor et al., 2016). 목표 염기에 대한 탈아민 작용이 이루어지지 않은 반대편 DNA 가닥에 nick이 생기면 이를 수선하는 mismatch repair (MMR) 시스템이 작동하여 상보적인 염기로 수선하여 염기교정 효율을 더욱 높일 수 있을 것으로 예상하였고, 탈아민화 염기의 반대 가닥에 nick을 만드는 nCas9 (D10A)를 사용하여 APOBEC1-XTEN-nCas9 (D10A)으로 구성된 BE3를 개발하였다 (Fig. 3). 그 결과 최대 37%까지 염기교정 효율이 증가하였다(Komor et al., 2016).
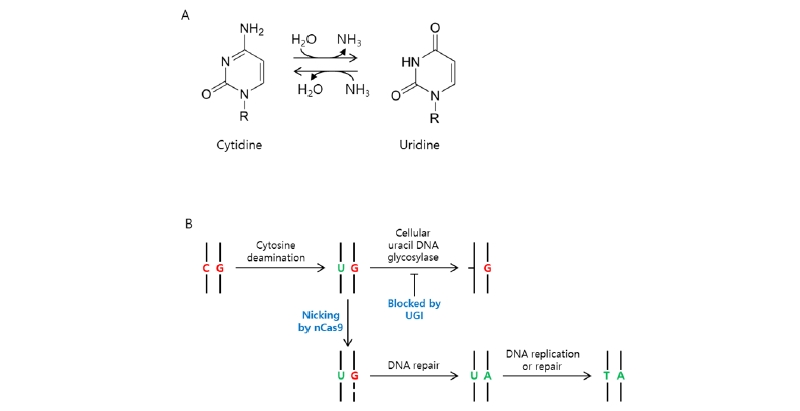
Fig. 3 iagrams of chemical responses and process of cytosine base editing in cell. (A) The diagram of chemical represents responses caused by deamination of cytidine. The deamination of cytidine results in uridine. (B) The diagram of consecutive responses started by cytosine deamination and resulted in transition from cytosine to thymidine in living cell.
Nishida et al. (2016)은 cytidine deaminase로서 인간의 AID의 상동유전자인 칠성장어 유래의 PmCDA1과 UGI를 BE3와 달리 nCas9 (D10A)의 카르복실 말단에 결합시킨 nCas9 (D10A)-PmCDA1-UGI를 개발하였다(Fig. 4). BE3에 비하여 염기교정 위치가 protospacer adjacent motif (PAM)에서 멀어진 20 bp 떨어진 위치이며, 교정범위도 6 bp로 일부 증가하였다(Fig. 4). 포유류 세포인 chinese hamster ovary (CHO) 세포에서 5.1 - 11%의 cytosine 염기교정 효율을 나타내었다(Nishida et al., 2016).
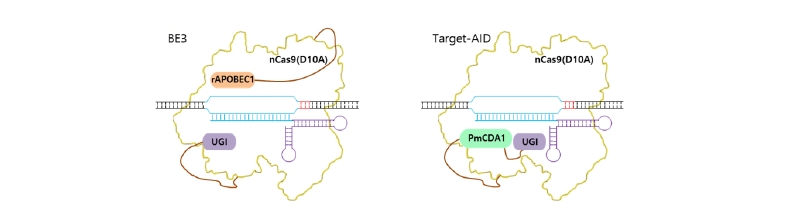
Fig. 4 Schematic comparison between two different cytosine base editors. There are two major cytosine base editors called BE3 and target- activation-induced cytidine deaminase (AID) using different cytosine deaminases developed independently by two research groups. Left panel is BE3 which consists of rAPOBEC1 fused to N-terminal of nCas9 (D10A) and UGI fused to C-terminal of nCas9 (D10A). Right panel is target-AID which consists of PmCDA1 and UGI fused to C-terminal of nCas9 (D10A).
BE3는 메틸화가 많이 되어있는 유전자나 CpG 염기서열에서 염기교정 효율이 낮은 문제점을 가지고 있으며, 이를 극복하기 위하여 메틸화 지역의 cytosine 염기교정 효율이 높은 cytidine deaminase를 탐색한 결과 인간 APOBEC3A는 메틸화가 집적된 유전자와 CpG 염기서열의 cytosine을 교정할 수 있는 능력이 있음을 증명하였다(Wang et al., 2018). 인간세포용 CBE가 개발된 이후 작물에서는 Zong et al. (2017)이 cytosine base editor, BE3 운반체인 APOBEC1-XTEN-Cas9 (D10A)-UGI를 작물용으로 코돈을 변화시키고, 옥수수 Ubiquitin-1 (Ubi-1) 프로모터로 발현을 시키는 작물용 cytosine base editor를 개발하여 벼, 밀 그리고 옥수수에서 CBE가 작동함을 최초로 증명하였다. Lu와 Li 등은 동일한 cytosine base editor를 이용하여 벼 및 토마토에서 염기교정을 독자적으로 보고하였다(Li et al., 2017; Lu and Zhu 2017). Shimatani et al. (2017)은 벼의 코돈에 최적화된 nCas9-PmCDA를 벼 캘러스에 형질전환하여 18.3%의 높은 염기교정 효율을 얻었다. 또한 토마토에서 염기교정을 확인하기 위하여 애기장대의 코돈에 최적화된 nCas9과 PmCDA1을 운반체로 하여 Della와 ETR1유전자에 대한 염기교정을 확인하였다. 쥐 유래 cytosine deaminase인 APOBEC1 기반의 BE3는 5 bp의 좁은 교정 범위와 GC 염기서열에 대한 교정 효율이 낮아, 인간 APOBEC3A (A3A-PBE)를 사용한 결과 밀, 보리, 감자에서 BE3보다 훨씬 높은 효율을 보였으며, 17 bp의 매우 넓은 교정 범위와 CpG 반복서열 등에서 염기교정 효율이 낮아지지 않았다(Zong et al., 2018). 이상의 연구결과를 통하여 작물에서 cytosine base editor가 성공적으로 활용될 수 있음이 증명되었다.
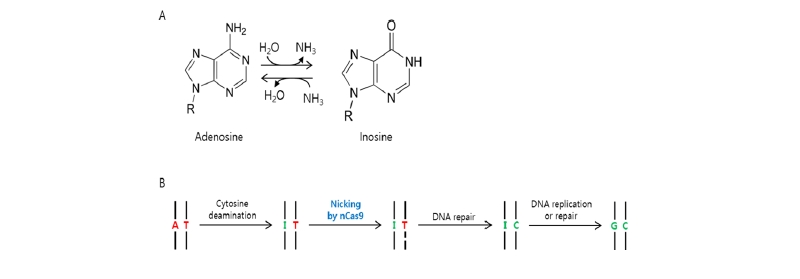
Fig. 5 Diagrams of chemical response and functional processes mechanisms of adenosine base editing in cell. (A) The diagram represents chemical response caused by deamination of adenosine. The deamination of adenosine results in inosine. (B) The diagram of consecutive responses started by adenosine deamination and resulted in transition from adenosine to guanidine in living cell.
Adenine base editor (ABE)의 개발 및 식물용 ABE의 개발
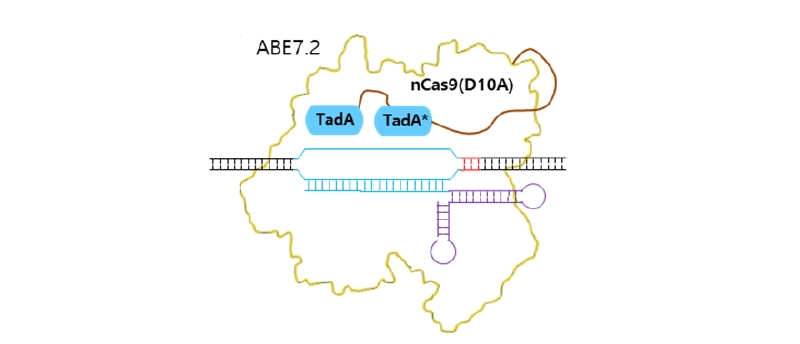
Adenine의 아민기를 제거하면 inosine이 만들어지고 일련의 세포 내 DNA 수선기작에 의해서 최종적으로는 guanine으로 치환된다(Fig. 5). 생물체에서 DNA의 adenine을 탈아민화 시키는 효소는 현재까지 알려져 있지 않으므로, Gaudelli et al (2017)은 RNA adenosine deaminase인 대장균의 TadA, 인간 ADAR2, 생쥐 ADA 그리고 인간 ADAT2를 nCas9에 결합하여 탈아민 활성을 측정하였으나, DNA의 adenine 탈아민 활성을 나타내지는 않았다. 따라서, tRNA adenosine deaminase인 대장균의 TadA를 DNA adenine deaminase로 인공 진화시켜, DNA adenine deaminase 활성을 갖는 TadA 돌연변이를 찾고, 돌연변이를 지속적으로 축적하여 TadA 단백질 중 14 개의 아미노산이 변화된 TadA* 돌연변이를 최종적으로 선발하였다(Gaudelli et al., 2017). TadA* 돌연변이는 대장균에서는 매우 높은 DNA adenosine deaminase 효율을 보였으나 포유류 세포에서는 그 효율이 매우 낮았다. 이를 극복하기 위하여 TadA 단백질은 동형이량체 (homodimer)로서 작동하기 때문에 TadA*과 야생형 TadA를 결합하여 이량체를 형성할 수 있도록 nCas9에 결합시킨 TadA-TadA*-nCas9을 개발하여 ABE7.10이라고 명명하였다(Fig. 6). ABE7.10은 동물세포에서 53% 의 교정 효율과 4 - 7 bp의 교정범위를 가지며, off target 비율은 0.1% 미만의 매우 효율적인 시스템으로 증명되었다(Gaudelli et al., 2017).
네 개의 연구그룹이 ABE를 식물에 적용가능한지 검토하였다. Kang et al. (2018)은 식물 코돈에 최적화된 TadA*를 nCas9에 결합시켜 애기장대와 유채의 염기교정에 성공하였다 (Kang et al., 2018). Li et al. (2017)은 벼와 밀에서 다양한 조합의 adenine 염기교정 운반체를 사용하여 최적 조건을 확인하였다. TadA-TadA*은 nCas9의 아미노기에 결합하고 3 개의 nuclear localization signal (NLS)가 nCas9의 카르복실말단에 결합되어있을 때 가장 효율이 높았으며, esgRNA (escorted single CRISPR-Cas9 guide RNA)는 sgRNA보다 높은 효율을 보였다 (Li et al., 2017; Li et al., 2018). Yan et al. (2018)은 벼 코돈에 최적화된 TadA7.10을 제작하고 벼에서 ABE가 성공적으로 작동함을 증명하였다. Hua et al. (2018)은 TadA7.10을 SpCas9와 SaCas에 결합하고 VirD2 NLS를 사용하여 벼에서 성공적으로 목표 유전자의 염기교정을 수행하였다.
PAM 염기서열 및 염기교정 범위의 확장
염기교정 시스템은 일반적으로 매우 좁은 염기교정 범위(4 - 5 bp)를 갖고 있으며, CRISPR 핵산분해효소가 작동하기 위해서는 목표 염기서열에 PAM가 필요하다. 따라서 특정 PAM 서열 및 좁은 염기교정 범위로 인하여 염기교정 가능 부위는 매우 제한적이게 된다. 이를 극복하기 위한 방법의 하나로 다양한 미생물로부터 서로 다른 PAM 서열을 갖는 Cas 핵산분해효소인 Cpf1, SaCas9 등을 분리하였다(Ran et al., 2015; Zetsche et al., 2015). 또 다른 방법으로 Cas 핵산분해효소에 돌연변이를 유도하여 다양한 PAM 서열을 인식하도록 만드는 방법이 개발되었다. Kleinstiver et al. (2015a; 2015b)은 SaCas9 과 SpCas9에 다양한 돌연변이를 유도하여 서로 다른 PAM을 인식할 수 있도록 제작하였다. 그러나, 여전히 PAM 염기서열에 대한 더 많은 확장성이 요구되어 단백질 공학을 이용하여 NG를 PAM으로 인식하는 Cas9 돌연변이를 제작하였다(Hu et al., 2018; Nishimasu et al., 2018).
Hu et al. (2018)은 phage-assisted continuous evolution (PACE)를 이용하여 PAM 염기서열이 확장된 Cas9 변이체를 확보하여 xCas9으로 명명하였으며 기존의 PAM 서열인 NGG가 확장된 NG, GAA, GAT 서열을 PAM으로 인식한다. 또한 xCas9은 PAM 염기서열의 확장에도 불구하고 Cas9보다 DNA 특이성은 증가하였다고 보고하였다.
Nishimasu et al. (2018)은 단백질구조에 기반한 단백질공학으로 PAM 염기서열이 확장된 Cas9-NG를 개발하였다. Cas9-NG는 NGA, NGT, NGG, 그리고 아주 낮은 비율로 NGC를 인식한다. 현재까지 염기교정에 사용되는 다양한 Cas9과 이들이 인식하는 PAM 서열들은 Table 1과 같다.
|
Table 1. Cas9 derivatives and their protospacer adjacent motif (PAM) sequences used in base editors. 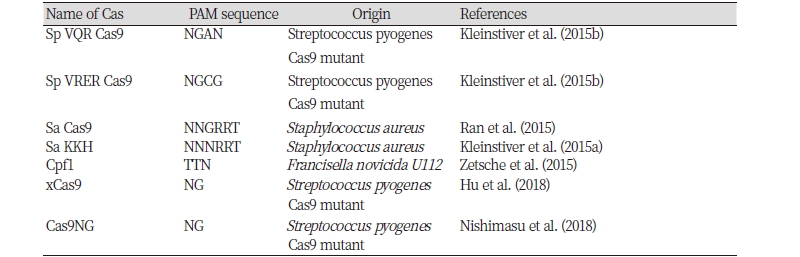
|
|
N = A, T, G, C. |
일반적으로 ABE와 CBE의 염기교정 범위가 4 - 5 bp인데 용도에 따라 이를 확장 혹은 좁히기 위한 노력이 이루어 졌다. CBE의 염기교정 범위 확대를 위하여 GCN4 펩타이드를 10번 반복하여 nCas9 (D10A)에 결합시키고, GCN4와 상호작용하는 항체인 ScFV를 APOBEC1-UGI와 결합하여 APOBEC1의 효소 활성 범위를 4 - 16 bp까지 확장시켰다. 이를 base editor for programming larger C to U scope (BE-PLUS) 라고 명명하였다. 반대로 염기교정의 엄밀성을 높이기 위하여 APOBEC1에 돌연변이를 유발하여 염기교정 범위를 1 - 2 bp로 좁힌 결과도 보고되었다(Kim et al., 2017).
동물세포에서 PAM 염기서열 확장에 성공한 이 후 다양한 연구그룹이 xCas9과 Cas9-NG를 이용한 ABE와 CBE 운반체를 작물에 적용하기 시작하였다. Cas9-NGv1에 CBE 또는 ABE를 적용하여 벼와 애기장대에서 PAM 염기서열이 확장된 염기교정을 유도하였다(Endo et al., 2019; Negishi et al., 2019). Ren et al. (2019)은 Cas9-NG는 벼에서 NGG, NAC, NTG, NTT와 NCG을 PAM으로 인식할 수 있을 뿐만 아니라 NAC, NTG, NTT, NCG도 PAM으로 인식할 수 있다고 확인하였다.
확장된 PAM 서열을 갖는 xCas9과 Cas9-NG를 이용하여 염기교정시스템을 제작하고 효율을 비교하였다. 동물세포에서와 달리 식물에서 xCas9은 NGG에서 특이적으로 활성을 보이고, NGH (H = A, C, T)에서는 매우 낮은 효율을 보였다. 반면에 Cas9-NG는 동물세포에서와 마찬가지로 식물에서도 NGG PAM에 대한 인식 효율이 매우 낮고, NG PAM에서는 높은 인식 효율을 보였다 (Zhong et al. 2019). Hua et al. (2019a)은 xCas9은 벼에서 NGG, NGT, NGA에서 indel 돌연변이를 일으키는 것을 확인하였으나, 염기교정 시스템을 결합시킨 xCas9은 염기교정을 하지 못한다고 보고하였다. 반면에 염기교정시스템이 결합된 Cas9NG는 NGH에서 PAM을 인식하여 염기교정을 수행함을 확인하였다(Hua et al., 2019a).
동물시스템에서 개발된 다양한 종류의 PAM을 인식하는 SpCas9 과 SaCas9의 변이체에 ABE와 CBE를 결합하여 다양한 운반체를 개발하고 벼에서 모두 성공적으로 염기교정이 이루어짐을 증명하여 염기교정 시스템의 적용성을 확장하였다(Hua et al., 2019b; Qin et al., 2019).
Base editor를 이용한 작물 염기교정 사례 및 전망
작물에서 최초의 염기교정 사례는 CBE를 활용하여 벼에서 OsCDC48, OsNRT1.1B 그리고 OsSPL14 세 개의 유전자를 동시에 교정하고, 밀에서는 LOX2를 옥수수에서는 ZmCENH3를 교정한 것이었다(Zong et al., 2017). CBE를 사용하여 SLR1의 TVHYNP 도메인에 바로 인접한 cytosine을 threonine으로 교정하여 T1 식물체에서 키가 작아지는 표현형을 관찰하였으며, 질소 운반체인 NRT1.1B 의 327 번째 threonine을 methionine으로 교정하였으나 표현형에 대한 보고는 없었다 (Lu and Zhu, 2017). Li et al. (2017)은 CBE로 PHYTOENE DESATURASE (OsPDS)와 전분 합성 유전자인 STARCH BRANCHING ENZYME IIb (OsSBEIIb)를 염기교정 하였다고 보고하였으나, 표현형을 보고하지는 않았다.
Shimatani et al (2017)은 벼에서 ACETOLACTATE SYNTHASE (ALS) 유전자의 96 번째 alanine을 valine으로 교정하여 imzamox 제초제에 저항성인 벼를 개발하였다. 또한 FTP1e와 ALS를 동시에 염기 교정하여 약 85%의 캘러스가 두 곳에서 모두 염기교정에 성공하였음을 확인하였다. 또한, 토마토의 Della와 ETR 유전자를 교정하는데 성공하였다고 보고하였다. 17 bp 의 넓은 염기교정 범위를 갖는 인간 APOBEC3A를 이용하여 밀의 TaALS, TaMTL, 벼의 OsCDC48, OsNRT1.1B, 감자의 StGBSS를 효과적으로 염기 교정하였다. 따라서, APOBEC3A는 작물에서도 넓은 염기교정 범위를 갖는 효과적인 시스템으로 사용될 수 있음을 증명하였다(Zong et al. 2018).
ABE를 사용하여 최초로 애기장대와 유채의 FT 유전자와 PDS 유전자에 염기교정을 수행하여 개화지연과 알비노 식물체를 확보할 수 있었다(Kang et al., 2018). 지질 생합성의 중요한 효소인 Acetyl-coenzyme A carboxylase (OsACC)의 변이는 phenylpyrazoline, cyclohexanedione, phenylpyraxoline 화합물계열의 제초제에 대한 저항성을 부여한다. Li et al. (2018)은 OsACC 유전자의 C2186R 변이를 만들기 위하여 염기교정을 수행하여 haloxyfop-R-methyl에 대한 저항성 벼를 제작하는데 성공하였다. 이후 다양한 그룹에서 ABE를 사용하여 염기교정에 성공하였다고 보고하였다. Yan et al. (2018)은 벼의 OsSERK2, OsWRKY45, OsMPK6유전자를, Hua et al. (2018)은 벼의 OsSLR1, OsSPL14, OsSPL17, OsSPL16, OsSPL18 유전자를 염기 교정하는데 성공하였다. 그러나 교정 식물체의 표현형에 대한 보고는 없었다. 또한 확장된 PAM 인식도구를 유전자 편집도구에 결합하여 유전자교정에 성공한 사례가 보고되었다. Cas9-NG에 결합된 CBE를 사용하여 ALS 와 DROOPING LEAF (DL) 유전자의 편집에 성공하였다 (Yan et al. 2018). Ren et al (2019)는 다양한 PAM 인식 Cas9 결합 ABE와 CBE를 사용하여 OsGSK4, OsMPK11, OsMPK7, OsMPK10, OsMPK8, OsSERK1, OsSERK2, OsETR2, OsBZR1을 단일 유전자 혹은 여러 유전자를 동시에 염기교정 하여 염기교정도구의 사용 범위가 확장되었음을 증명하였다(Ren et al. 2019). Hua et al. (2018)은 OsmiRNA156이 결합하는 OsSPL13 ~ 18까지 6개 유전자를 염기교정 하였다.
염기교정기술의 농업적 활용 전망
동물에서 개발된 염기교정기술이 최근에 식물에의 활용 시스템이 확립되고, 모델작물의 기능이 잘 알려진 유전자를 중심으로 활성을 검정하는 단계를 완료하였다. 이후 작물의 정밀 육종분야에 본 기술을 활용하기 위한 노력들이 다양한 연구 그룹에서 시작하고 있지만 아직 초기 단계이다. 따라서, 실질적인 실용화 사례들은 아직까지 보고되지 않았지만, CRISPR/Cas9과 비교하였을 때 본 기술은 기능이 이미 알려진 유전자의 성능을 향상시키는 분야에서 매우 유용하게 활용될 것으로 생각한다. 실험관 수준의 인위 점 돌연변이와 일시 발현기술을 활용한 유전자기능 향상을 검정하여 정확한 유전자 교정 목표를 정한 뒤 작물에서 원하는 부위의 염기서열을 교정할 수 있을 것이다. 또한, 농업형질에 중요한 자연적 SNP 돌연변이 특성을 분석하고 변이된 염기서열을 선발한다면, 본 기술을 이용하여 우수품종에 목적하는 SNP를 인위적으로 도입하여 육종효율을 증진시킬 수 있을 것으로 생각한다.
Conclusion
2016년 cytosine base editor가 동물에서 최초로 개발된 이래로 3년만에 주요 작물에서 위와 같이 다양한 적용사례들이 보고가 되었다. 이것은 염기교정기술의 중요성, 파급성 및 사용의 편이성을 나타내고 있는 것이다. 본 고에서 기술한 바와 같이 염기교정은 상용화가 가능한 수준으로 기술 발전이 이루어져 있으며 유전자교정 작물 규제의 SDN-1에 해당하는 기술로서 향후 규제를 회피할 가능성이 높아 작물 실용화를 위한 유망 기술로 생각된다. 또한, 일반적인 Cas9에 비하여 1bp의 원하는 염기서열만 교체한다는 점에서 훨씬 정교한 유전자교정이 가능하여 정밀 육종의 시대를 열어갈 기술로 생각된다. 따라서, 본 기술을 활용하여 개발된 작물에 대한 소비자 및 사회적인 거부감도 적을 것으로 기대되어 생명공학 작물의 상용화를 위한 좀 더 진보된 기술로 이 분야에 대한 연구 및 투자가 이루어져야 할 것으로 생각한다.
Authors Information
Rigyeong Kim, National Institute of Agricultural Sciences, Professional Technician
Jaeeun Song, National Institute of Agricultural Sciences, Technician
Eunji Ga, National Institute of Agricultural Sciences, Master course student
Myung Ki Min, National Institute of Agricultural Sciences, Post-doctoral fellow
Jong-Yeol Lee, National Institute of Agricultural Sciences, Researcher
Sun-Hyung Lim, National Institute of Agricultural Sciences, Researcher
Beom-Gi Kim, National Institute of Agricultural Sciences, Senior Researcher