Introduction
국내의 육류 소비량은 2010년 431,299톤에서 2019년 672,000톤으로 증가하였고, 1인당 육류 소비량 또한 2010년 8.83 kg에서 2019년 13 kg으로 매년 꾸준히 증가하는 추세이다(KMTA, 2021). 한우와 수입육의 육질에 대한 소비자 조사결과 안정성, 신선도 그리고 근내지방도 부분에서 한우가 우수한 것으로 나타나 충분한 경쟁력을 가지고 있다고 보고되었으며(Choi et al., 2017), 이에 따라 한우 산업은 소비자의 수요를 충족시키기 위해 육질 관련 형질에 대한 지속적인 개량이 이루어져 왔다. 특히, 한우 산업 중 육용우로 많이 사용되는 거세우의 경우 육질등급 중 1등급이상(1++, 1+포함) 출현율이 2009년 57.2%에서 2019년 88.8%로 증가한 것을 통해 한우에 대한 개량이 성공적으로 이루어지고 있다고 판단된다.
가축의 경제형질에 대한 유전적 개량은 선발을 통해 이루어져 왔으며(Dekkers and Hospital, 2002), 한우의 경우 유전적 개량을 위해 유전능력평가를 실시하여 추정된 결과를 종축 선발의 참고자료로 활용해 왔다. 유전능력평가 방법 중 Henderson의 BLUP (best linear unbiased prediction)을 이용한 평가는 기존에 표현형정보를 근거로 종축을 선발하던 방식에서 표현형정보에 영향을 주는 나이, 분만계절, 성별 등 여러 환경적 요인을 효과적으로 보정하고 개체의 혈통정보를 활용하여 추정육종가(EBV, estimated breeding value)를 계산하는 방법으로 가축의 개량에 큰 영향을 주었다(Parnell, 1984). 그러나, BLUP을 이용한 유전능력평가는 혈통정보가 없거나 미흡한 개체의 경우 혈연적 연결점이 없어 EBV에 대한 정확도 추정이 불가능하고 같은 부모를 가진 형매 간의 유전적 변이를 계산하지 못해 동일한 EBV 추정이 이루어져 개체의 동시 선발이 일어나 정확한 유전능력평가의 어려움을 갖는다고 보고되었다(Shin, 2008, Heffner et al., 2009).
Meuwissen 등(2001)이 제시한 유전체 선발은 개체별로 유전체 전반에 분포하고 있는 수많은 마커에서 확보한 유전체정보와 표현형정보를 활용하여 유전능력을 추정하는 방법으로 혈통정보를 활용한 pedigree BLUP (PBLUP)보다 높은 정확도 추정이 가능하다고 보고되었다. 축산 선진국에서는 유전체 선발방법 중 genomic BLUP (GBLUP)과 Bayesian의 통계모델을 사용하여 유전체육종가(GEBV, genomic estimated breeding value)를 추정하는 연구가 추가적으로 보고되었으며(VanRaden et al., 2009, Rolf et al., 2010, Su et al., 2012), 국내 연구에서도 한우를 대상으로 EBV와 GEBV의 정확도를 비교 분석하는 연구가 활발히 진행되고 있다(Lee et al., 2013, Shin et al., 2018). 따라서, 본 연구는 PBLUP과 GBLUP으로 추정된 육종가와 정확도를 비교 분석하기 위해 거세우의 혈통 및 유전체정보를 활용하여 도체형질에 대한 육종가와 정확도를 추정하였다.
Materials and Methods
분석집단과 표현형정보
본 연구에서 사용된 검정집단은 경남지역 A축협 생축장에서 거세우 46두의 혈액, 개체이력번호 및 도체성적을 포함한 표현형정보를 제공받아 분석에 사용하였다. 축산물품질평가원과 (사)한국종축개량협회에서 개체이력번호를 조회하여 성별, 생년월일, 혈통정보 등을 수집하였다.
검정집단의 육종가를 추정하기 위해 사용된 분석방법은 혈통정보를 활용하여 EBV를 추정하는 PBLUP과 유전체정보를 활용하여 GEBV를 추정하는 GBLUP이며, 각 분석방법은 검정집단과 참조집단이 보유한 정보(혈통 및 유전체정보)를 바탕으로 개체 간의 혈연계수를 추정하고 표현형정보와 함께 육종가를 추정한다. 두 분석방법을 사용하기 위해서는 참조집단이 필요하다. 사용된 참조집단은 2가지로 참조집단Ⅰ은 혈통 및 표현형정보를 보유한 545,483두이며, Kim 등(2020)이 사용한 참조집단과 동일한 집단으로 본 연구에 사용하였다. 참조집단Ⅱ는 유전체 및 표현형정보를 보유한 16,972두이다. 검정집단과 참조집단Ⅰ, Ⅱ가 보유한 정보에 대해 Table 1에 나타냈다. 검정집단과 참조집단Ⅰ, Ⅱ가 공통으로 보유한 표현형정보는 농림축산식품부에서 고시한 제2014-4호 축산물등급판정 세부기준에 따라 도축 후 24시간 냉장 후 도체형질을 측정하였다. 도체중은 좌우 냉도체 중량의 합으로 측정하였고, 등심단면적은 좌우 흉추와 제 1요추 사이를 척추골과 직각으로 절개하여 최후 흉추쪽의 면적을 면적자로 측정하였다. 등지방두께는 등심단면적의 오른쪽면을 따라 복부쪽으로 3분의 2정도 들어간 지점을 측정하였으며, 근내지방도는 등심단면적 측정부위에서 근육 내 지방 침착도를 기준표(1 = devoid and 9 = abundant)와 비교하여 육안으로 측정하였다.
PBLUP으로 EBV 추정
PBLUP의 경우 혈통정보를 활용하여 개체 간의 혈연관계도(NRM, numeric relationship matrix)를 구축하기 위해 정확한 혈통정보가 필요하다. 혈통정보를 수집하기 위해 검정집단과 참조집단Ⅰ의 개체이력번호를 (사)한국종축개량협회에서 혈통정보 조회를 통해 3대까지 역추적하여 가계도를 구축하였다. 분석의 용이성을 위해 Animal, Sire 및 Dam으로 정렬하고, Pedigree viewer (The University of New England, New England, Australia)를 이용하여 혈통구조 및 이력번호 재배열을 실시하였다(Kinghorn and Kinghorn, 2010). 이후, 대용량 정보처리에 적합한 R (R Foundation for Statistical Computing, Vienna, Austria)을 사용하여 중복개체 및 불일치를 수정하였고, 확보된 검정집단과 참조집단Ⅰ의 가계도를 합쳐 NRM을 구축하여 EBV 추정에 사용하였다.
혈통정보를 활용하여 구축된 NRM을 BLUPF90 (Misztal et al., 2018)의 single trait animal model을 사용하여 형질 별 EBV, 예측오차편차(PEV, prediction error variance), 유전모수를 추정하였다. 고정효과는 출생 연도, 출생 월, 도축일령 등을 사용하였고, 혼합모형방정식은 아래와 같다(Henderson, 1975).
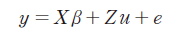 (1)
(1)
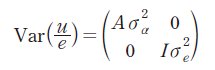 (2)
(2)
여기서, y는 도체형질의 관측치에 대한 벡터, X는 고정효과인 출생 연도, 출생 월, 도축일령 등에 대한 벡터, β는 고정효과에 대한 추정치의 벡터, Z는 개체에 대한 임의효과의 벡터, u는 개체에 대한 추정치의 벡터, e는 임의 오차의 벡터이고, , , 으로 가정하여 가 된다. 여기서, A는 NRM이고 는 상가적 유전분산, 는 임의 환경분산이다. 유전력은 로 h2은 추정된 유전력,은 유전분산, 은 잔차분산으로 전체분산 에서 유전분산이 차지하는 비율로 계산하였다.
GBLUP으로 GEBV 추정
GBLUP은 유전체정보를 활용하여 개체 간의 유전체혈연관계도(GRM, genomic relationship matrix)를 구축하고 GEBV를 추정한다. 분석에 사용될 유전체정보를 수집하기 위해 검정 및 참조집단Ⅱ의 모근 및 혈액에서 추출한 고순도 genomic DNA를 확보하였고, Bovine 50K SNP BeadChip Ver.2, Ver.3 및 Hanwoo 50K SNP BeadChip (Illumina Inc., San Diego, CA, USA)을 사용하여 유전체정보를 수집하였다. 수집된 대용량 유전체정보는 GenomeStudio (Illumina Inc., San Diego, CA, USA)를 사용하여 Plink 1.9 (Purcell et al., 2007)에 적합한 형식으로 변환시켰다. 이후, 검정집단과 참조집단Ⅱ의 유전체정보를 공통된 SNP 마커정보를 합쳐 47,450개의 SNP 마커를 확보하였고, Plink 1.9 program의 quality control 옵션인 MAF (minor allele frequency) 1% 미만, missing genotype 10% 이상, HWE (Hardy-Weinberg equilibrium) 10-4 미만을 사용하여 각각 2,493, 1,106, 8,942개의 SNP 마커를 제거하였다. 최종적으로 34,909개의 SNP 마커로 GRM을 구축하여 GEBV 추정에 사용하였다.
GBLUP은 PBLUP과 동일한 고정효과를 이용하였으나, 개체 간의 혈연적 관계계수를 가지고 있는 NRM 대신 유전체정보를 활용하여 구축한 GRM을 대입하여 GEBV 추정을 수행하였다. 분석에 사용된 혼합모형방정식은 기존과 동일하며 아래와 같다.
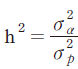 (3)
(3)
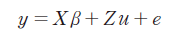 (4)
(4)
여기서, y는 도체형질의 관측치에 대한 벡터, X는 고정효과인 출생 연도, 출생 월, 도축일령 등에 대한 벡터, β는 고정효과에 대한 추정치의 벡터, Z는 개체에 대한 임의효과의 벡터, u는 개체에 대한 추정치의 벡터, e는 임의 오차의 벡터이고, , , 으로 가정하여 가 된다. 여기서, G는 GRM이고 는 상가적 유전분산, 는 임의 환경분산이다. 유전력은 PBLUP에서 사용한 동일한 방법으로 계산하였다.
정확도 추정
PBLUP과 GBLUP에서 나온 PEV를 아래와 같은 방정식을 사용하여 육종가의 정확도를 추정하였다.
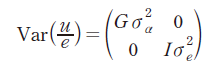 (5)
(5)
여기서, Acc는 추정된 육종가의 정확도, PEV는 추정된 육종가의 예측오차분산, σα2는 상가적 유전분산이다. PEV는 개체마다 추정된 육종가의 오류 편차 범위이며, 각 분석방법의 혼합모형방정식에 대한 역행렬계수로 모든 개체에서 쉽게 얻을 수 있고 는 BLUPF90 패키지의 REMLF90으로 계산할 수 있다.
추정된 육종가와 표현형 값의 상관계수 추정
검정집단의 표현형 값과 PBLUP과 GBLUP으로 추정된 육종가를 상관계수 추정 분석을 실시하였다. 추정방법은 R = Cor(G)EBV, Pheno로 R은 두 값의 Pearson`s product-moment 상관계수이며, 상관계수의 유의성 검증 결과인 p-value를 추정하여 육종가와 표현형 값의 유의성을 검증하였다. 상관계수 추정 방법은 PBLUP과 GBLUP에서 추정된 개체별 육종가가 표현형 값에 얼마나 근접하게 가지는 것을 확인하는 방법으로 상관계수 값과 방향성을 이용하여 분석방법의 정확성을 확인할 수 있다.
Results and Discussion
검정집단과 참조집단의 기초통계량
PBLUP에 사용한 참조집단Ⅰ과 GBLUP에 사용한 참조집단Ⅱ의 경우 평균 30개월령에 도축된 개체이고 검정집단의 경우 평균 29개월에 도축된 개체로 각각 구성하였으며, 이들의 기초통계량을 Table 2에 나타내었다. 각 집단의 도체중, 등심단면적, 등지방두께, 근내지방도의 평균과 표준편차는 검정집단에서 446 ± 43.6 kg, 98.3 ± 13.6 cm2, 14 ± 4.6 mm, 6.6 ± 1.7점으로 나타났고 참조집단Ⅰ에서 431.1 ± 43 kg, 91.1 ± 9.7 cm2, 13.2 ± 4.5 mm, 5.7 ± 1.9점, 참조집단Ⅱ에서 439.9 ± 51.3 kg, 94.8 ± 12.0 cm2, 14.2 ± 4.9 mm, 6.0 ± 1.9점으로 나타났다. 참조집단Ⅰ은 Kim 등(2020)이 사용한 집단과 같음으로 동일한 기초통계량을 가진다. Do 등(2016)이 보고한 내용에 따르면 한우사업단에서 수집한 집단의 도체중, 등심단면적, 등지방두께, 근내지방도가 357.1 ± 61.8 kg, 81.8 ± 11.5 cm2, 11.2 ± 5 mm, 4.24 ± 2.13점으로 본 연구결과보다 낮게 나타났는데, Lee 등(2020)은 통합자료를 구성하는 수소, 암소 및 거세우의 비율에서 나타나는 차이와 도축일령의 차이로 분석에 사용하는 집단의 기초통계량 차이가 나타날 수 있다고 보고하였다. 2019년 축산물통계연보에서 거세우의 도체중, 등심단면적, 등지방두께, 근내지방도가 445.8 kg, 95 cm2, 13.8 mm, 5.9점으로 보고하였고(KIAPQE, 2020), Lee 등(2020)은 강원도지역 사육농가에서 수집한 집단의 도체중, 등심단면적, 등지방두께, 근내지방도가 431.77 ± 51.43 kg, 91.22 ± 10.75 cm2, 13.30 ± 5.14 mm, 5.66 ± 1.88점으로 보고하였는데, 두 선행연구는 본 연구결과와 유사하게 나타났다. 평균 30개월령에 출하하는 일반 사육농가 한우의 육종가를 추정하기 위해서는 검정 개체와 유사한 기초통계량을 가진 참조집단이 필요하며, 본 연구에 사용된 검정집단과 참조집단Ⅰ, Ⅱ는 일반 사육농가를 대상으로 육종가 추정에 사용하기 적합한 집단으로 판단된다.
Table 2. Descriptive statistics according to reference and test population.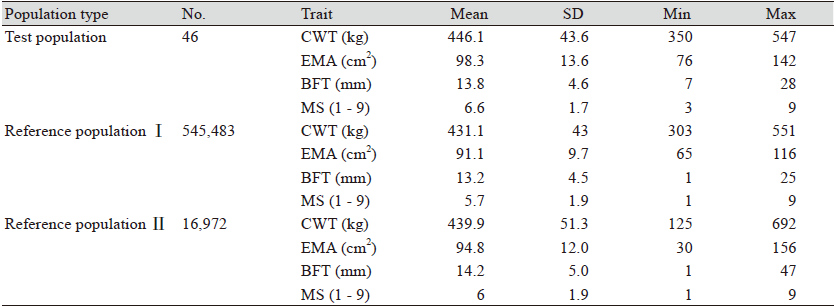
|
|
SD, standard deviation; CWT, carcass weight; EMA, eye muscle area; BFT, back-fat thickness; MS, marbling score. |
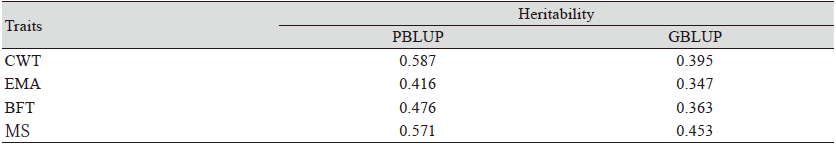
유전력 추정
검정집단에 대하여 참조집단Ⅰ의 혈통정보를 활용하는 PBLUP과 참조집단Ⅱ의 유전체정보를 활용하는 GBLUP으로 추정한 유전력을 Table 3에 나타냈다. 도체중, 등심단면적, 등지방두께, 근내지방도 순으로 PBLUP의 유전력은 0.587, 0.416, 0.476, 0.571이고 GBLUP의 유전력은 0.395, 0.347, 0.363, 0.453으로 두 분석방법에서 계산된 유전력은 중도에서 고도의 유전력을 가졌으며, 가장 높은 유전력을 가진 형질은 PBLUP에서 도체중, GBLUP에서 근내지방도로 나타났고 가장 낮은 유전력을 가진 형질은 PBLUP와 GBLUP에서 등심단면적으로 나타났다. PBLUP의 유전모수 추정에 관한 선행연구를 살펴보면, Kim 등(2020)이 보고한 도체형질의 유전력과 본 연구결과가 동일한데, 이는 동일한 참조집단을 사용하여 나타난 결과로 판단된다. Park 등(2020)은 칡소를 대상으로 도체중, 등심단면적, 등지방두께, 근내지방도에 대한 유전력 분석 결과를 각각 0.50, 0.37, 0.35, 0.53으로 보고하였으며, 본 연구결과보다 낮게 나타났다. Mehrban 등(2021)은 한우개량센터에서 생산된 한우를 대상으로 도체중, 등심단면적, 등지방두께, 근내지방도에 대한 유전력 분석 결과를 각각 0.38, 0.43, 0.50, 0.61로 보고하였으며, 도체중을 제외한 나머지 형질에서 본 연구와 유사하였다. GBLUP의 유전모수 추정에 관한 선행연구를 살펴보면, Shin 등(2018)은 한우 암소를 대상으로 도체중, 등심단면적, 등지방두께, 근내지방도에 대한 유전력 분석 결과를 0.520, 0.413, 0.451, 0.434로 보고하였으며, 근내지방도를 제외한 나머지 형질에서 본 연구결과보다 높게 나타났다. Utrera와 Van Vleck (2004)은 1962 - 2004년까지 출시한 72개의 bovine 논문을 조사하여 도체중, 등심단면적, 등지방두께, 근내지방도의 유전력이 각각 평균 0.40, 0.36, 0.40, 0.37로 보고하였으며, 본 연구결과 중 GBLUP으로 추정된 유전력과 유사하였다.
Cho (2013)와 Miar 등(2013)은 유전력 추정에 있어 분석에 사용되는 집단의 혈연계수, 크기 및 통계 분석방법 등으로 유전분산의 결과가 달라진다고 보고하였고, Lee (2012)는 PBLUP으로 추정한 유전력의 경우 혈통정보 기반으로 구축되는 NRM의 멘델리안 샘플링 효과가 전형매(full sibling)은 0.5, 반형매(half sibling)은 0.25로 추정된다고 보고하였다. 이를 통해 혈통구조에 따라 PBLUP의 혈연계수 평균값이 정해져 유전적 다양성을 확인하기 어렵다고 판단되며, 본 연구에서 추정된 PBLUP의 유전력은 중복된 혈연계수의 값이 증가함에 따라 추정해야 할 잔차분산 범위가 감소되어 유전력이 증가한 것으로 생각된다. 반면, GBLUP의 GRM은 NRM처럼 동일한 혈연계수의 평균값이 아닌 각 개체마다 가진 유전자형의 차이를 통해 추정된 혈연계수를 사용하기 때문에 중복된 혈연계수의 값이 PBLUP보다 적으며, 다양한 오차범위를 가짐에 따라 잔차분산의 값이 커져 유전력이 감소한 것으로 생각된다.
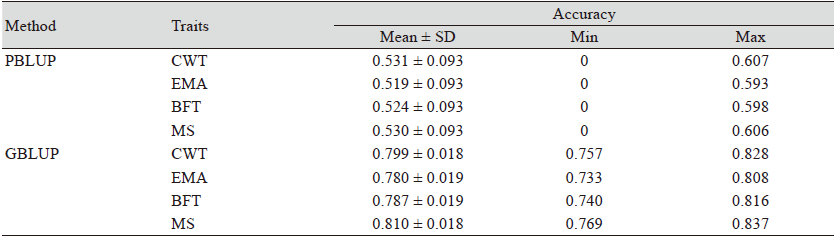
정확도 추정
검정집단에 대하여 참조집단Ⅰ의 혈통정보를 활용하는 PBLUP과 참조집단Ⅱ의 유전체정보를 활용하는 GBLUP으로 추정한 정확도를 Table 4에 나타냈다. 도체중, 등심단면적, 등지방두께, 근내지방도 순으로 PBLUP의 정확도는 0.531 ± 0.093, 0.519 ± 0.093, 0.524 ± 0.093, 0.530 ± 0.093이고 GBLUP의 정확도는 0.799 ± 0.018, 0.779 ± 0.019, 0.786 ± 0.019, 0.810 ± 0.018으로 나타났으며, PBLUP의 정확도보다 GBLUP의 정확도가 50.1 - 53.1% 높게 추정됨을 확인하였다. 정확도의 최솟값과 최댓값을 살펴보면, 최솟값의 경우 PBLUP은 모든 형질에서 정확도가 0으로 나타났는데, 참조집단과의 혈연계수가 낮아서 정확도 추정이 불가능하였고, GBLUP은 도체중, 등심단면적, 등지방두께, 근내지방도 순으로 0.757, 0.733, 0.740, 0.769로 모든 개체에서 정확도 추정이 가능하였다. 최댓값의 경우 도체중, 등심단면적, 등지방두께, 근내지방도 순으로 PBLUP은 0.607, 0.593, 0.598, 0.606, GBLUP은 0.828, 0.808, 0.816, 0.837로 차이가 평균 0.22로 나타났는데, 이는 검정집단과 참조집단Ⅰ의 혈통정보로 구축한 NRM의 혈연계수가 검정집단과 참조집단Ⅱ의 유전체정보로 구축한 GRM의 유전체혈연계수보다 낮게 추정되어 나타난 결과로 판단된다.
선행연구를 살펴보면, Shin 등(2018)은 일반 사육농가의 한우 암소를 대상으로 혈통 및 유전체정보를 활용하여 도체중, 등심단면적, 등지방두께, 근내지방도에 대한 정확도 분석을 실시하였고, 그 결과 PBLUP의 정확도는 0.441, 0.458, 0.464, 0.472, GBLUP의 정확도는 0.542, 0.509, 0.521, 0.513으로 보고하였으며, Lee 등(2013)은 후대검정우를 대상으로 혈통 및 유전체정보를 활용하여 도체중, 등심단면적, 등지방두께, 근내지방도의 정확도를 분석한 결과 PBLUP의 정확도는 0.6047, 0.6748, 0.6748, 0.7364이고 GBLUP의 정확도는 0.6176, 0.6812, 0.6812, 0.7416으로 보고하였다. 두 선행연구에서 혈통정보를 활용하여 정확도를 추정한 PBLUP보다 유전체정보를 활용하여 정확도를 추정한 GBLUP의 정확도가 높게 추정되었으며, 본 연구결과와 부합하였다. 하지만 Lee 등(2013)의 연구에서는 PBLUP와 GBLUP의 평균 정확도 차이가 Shin 등(2018)과 본 연구의 결과보다 작은 것을 확인하였는데, 분석에 사용된 후대검정우 집단의 혈통정보를 이용하여 구축한 혈연계수가 상당히 높아 PBLUP의 정확도가 높게 추정되었다고 보고하였다. 또한, Jang 등(2019)은 검정개체가 가지고 있는 후대개체의 수에 따라 정확도가 크게 바뀐다고 보고하였는데, 이는 일반 사육농가의 암소의 경우 연결되는 개체 수가 적어 혈통정보를 이용한 능력검정의 한계점으로 보인다. 따라서, Shin 등(2018)과 같이 일반 사육농가에서 혈통정보가 없거나 미흡한 개체의 경우 혈통정보를 활용하여 유전능력을 추정하는 PBLUP보다 동일한 SNP 마커를 활용하여 유전능력 추정이 가능한 GBLUP이 효과적인 능력평가방법이지만 혈연계수가 높은 개체의 경우 혈통정보를 이용한 PBLUP으로도 높은 정확도 추정이 가능할 것으로 사료된다.
표현형정보와 분석방법에 따라 추정된 육종가의 상관분석
본 연구에 사용된 상관분석은 PBLUP과 GBLUP으로 추정한 개체별 육종가와 표현형 값을 이용하여 상관관계를 추정하는 방법으로 높은 정의 상관관계를 가질수록 표현형에 근접하고 정확한 육종가 추정이라고 할 수 있다. 검정집단에 대하여 참조집단Ⅰ의 혈통정보를 활용하여 PBLUP으로 추정된 EBV와 참조집단Ⅱ의 유전체정보를 활용하여 GBLUP으로 추정한 GEBV를 검정집단의 표현형정보와 상관분석을 통해 개체마다 추정된 육종가가 표현형 값에 얼마나 근접하는지 확인하였고, 그 결과를 Fig. 1에 나타냈다.
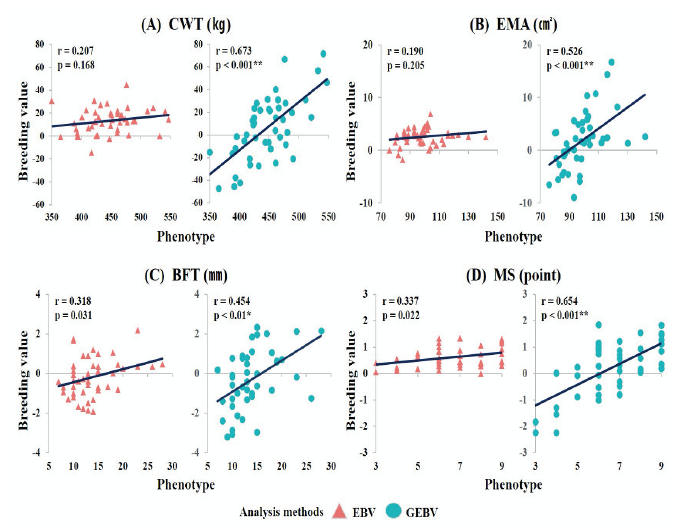
Fig. 1. Phenotype correlation with EBV and GEBV by trait according to analysis methods. CWT, carcass weight; EMA, eye muscle area; BFT, back-fat thickness; MS, marbling score; r, correlation coefficient; p, p-value; p < 0.05, significance; p < 0.01*, middle level significance; p <0.001**, high level significance; EBV, estimated breeding value using pedigree best linear unbiased prediction (PBLUP); GEBV, estimated breeding value using genomic best linear unbiased prediction (GBLUP).
Fig. 1의 PBLUP으로 추정된 EBV와 GBLUP으로 추정한 GEBV의 분포도를 살펴보면, 다양한 표현형 값을 나타내는 검정 개체들에 비해 EBV의 분포도가 상당히 좁은 것을 확인할 수 있으며, GEBV는 EBV보다 넒은 분포 범위를 나타내고 있다. 도체중, 등심단면적, 등지방두께, 근내지방도 순으로 EBV와 표현형 값의 상관계수는 0.207, 0.190, 0.318, 0.337로 나타났고, GEBV와 표현형 값의 상관계수는 0.673, 0.526, 0.454, 0.654로 모든 형질에서 EBV의 상관계수보다 GEBV의 상관계수가 높은 정의 상관관계로 나타났다. GEBV와 EBV에 대한 상관계수의 유의성 검증 결과를 살펴보면, GEBV의 p-value 값은 도체중, 등심단면적, 등지방두께에서 고도의 유의성(p < 0.001)을 보였고 등지방두께에서 중도의 유의성(p < 0.01)로 나타났으며, EBV의 p-value 값은 등지방두께와 근내지방도에서 낮은 유의성(p <0.05)을 보였지만 도체중과 등심단면적에서 유의적인 결과가 나타나지 않았다. 상관계수의 유의성 검증은 분석에 사용된 표본의 수가 많을수록 유의적인 결과 및 높은 정확도를 나타낸다. 그러나, 본 연구에서는 동일한 표본 수로 분석하였고, 그 결과 GEBV의 p-value 값이 고도의 유의성으로 높은 정확성을 가지며, 높은 정의 상관관계로 표현형 값에 가장 근접하게 추정되어 정확한 육종가 추정 방법이라고 판단된다. 개체의 표현형 값에 적합한 육종가를 추정하기 위해서는 분석에 사용되는 참조집단의 구성이 중요하다. Hayes와 Goddard (2008), Goddard (2009), Pszczola 등(2012)이 보고한 내용에 따르면, 참조집단의 유전적 조성의 변화에 따라 혈연계수, 유전자의 효과, SNP 마커효과 등에 큰 영향을 미치며, PBLUP과 GBLUP에 사용되는 참조집단의 경우 전형매 및 반형매 위주로 한쪽에 치우친 집단이 아닌 다양한 혈연적 연결점을 갖는 개체 및 세대를 추가하여 유전적 다양성을 확보하는 것이 중요하다. 따라서 한우를 대상으로 정확도 높은 육종가 추정을 위해서는 한우 집단에 적합한 참조집단 구성이 필요하다고 판단된다.
Conclusion
검정하고자 하는 개체의 적합한 육종가를 추정하기 위해서는 정확한 능력평가 방법이 필요하다. 본 연구는 혈통정보를 활용하여 EBV를 추정한 PBLUP과 유전체정보를 활용하여 GEBV를 추정한 GBLUP의 결과를 비교 분석하였다. 그 결과 GBLUP에서 추정한 정확도가 PBLUP으로 추정한 정확도에 비해 50.1 - 53.1% 높게 추정되었고, 검정집단의 표현형 값과 각 분석방법으로 추정된 육종가를 사용하여 상관관계를 분석한 결과 PBLUP에 비해 GBLUP이 높은 정의 상관관계를 가짐으로써 정확한 육종가 추정이 가능한 것으로 확인하였다. 향후 지속적인 PBLUP과 GBLUP 모델의 검증을 위해서는 다양한 혈연적 연결점을 갖는 개체 및 세대를 추가하여 유전적 다양성을 확보하는 것이 중요하며, 국내 한우 산업에 적합한 참조집단을 구성하는 것이 중요하다고 판단된다.
Authors Information
Eun Ho Kim, https://orcid.org/0000-0002-0882-8377
Du Won Sun, https://orcid.org/0000-0002-0181-5303
Ho Chan Kang, https://orcid.org/0000-0002-2948-5895
Ji Yeong Kim, https://orcid.org/0000-0001-8458-2789
Cheol Hyun Myung, https://orcid.org/0000-0001-8697-1657
Doo Ho Lee, https://orcid.org/0000-0002-2174-7897
Seung Hwan Lee, https://orcid.org/0000-0003-1508-4887
Hyun Tae Lim, https://orcid.org/0000-0001-6221-4942