Introduction
삶의 질 향상을 추구하는 사회 추세에도 불구하고 빈번하게 발생하는 식중독사고로 인하여 소비자의 건강한 먹거리에 대한 관심이 날로 증가하고 있다. 식중독을 일으키는 원인 물질은 수백 가지 이상이지만 대부분의 식중독 사고는 세균이나 바이러스에 오염된 식품 섭취로 인해 발생하는 것으로 알려져 있다(CDC, 2018). 농축산물 생산과정에서의 세균 오염은 주로 토양, 부숙되지 않은 퇴비, 지하수, 야생동물의 분변 등을 통해 발생하며, 가공 또는 유통과정에서는 세균에 오염된 시설, 기계, 그리고 용기의 표면과 세척수 등에 의한 교차 오염에 의해 발생한다(Beuchat, 2002).
농축산물의 생산과정에서 발생하는 세균 오염은 오염원이 다양하고 방대하여 근원적으로 해결하기가 매우 어렵다. 이보다는 상대적으로 관리가 쉬울 것으로 여겨지는 농식품 가공 시설에서 세균 오염 문제가 빈발하는 주된 원인 중 하나는 세균에 의해 생성되는 생물막(biofilm) 때문이다(Zhao et al., 2017). 생물막은 미생물 군락이 자기들끼리 또는 가공시설이나 농산물 표면 등에 단단하게 부착되어 있으면서 기질로 둘러싸여 고착된 것을 말한다(Donlan, 2002). 생물막은 미생물이 생성하여 분비하는 다당류 위주의 세포외 중합물질(EPS, extracellular polymeric substance)로 이루어져 있으며, 열악한 외부환경으로부터 미생물을 보호하고 외부로부터 영양분을 포획하여 공급하는 역할을 한다. 생물막에 부착된 미생물은 부유 미생물보다 항생제, 소독제, 고온, 빛, 건조 등에 더 잘 견디는 것으로 알려져 있다(Poulsen, 1999).
식품이 접촉되는 가공시설의 표면에 생성된 생물막을 확인하는 주된 방법은 검사자의 감각에 의존하여 무지갯빛을 띄는 부위 또는 미끈거리는 부위를 찾거나 면봉으로 표면의 시료를 채취한 다음 세균을 배양하는 것이다. 이러한 방법들은 검사자의 주관에 의존하기 때문에 부정확하거나 배양에 며칠 이상의 오랜 시간이 소요되는 단점이 있다. 또한, 생물막에 존재하는 세균은 움직이지 않기 때문에 ATP (adenosine triphosphate)를 매우 적게 생성하여 식품 위생 신속검사에 많이 사용되는 오염도 검사기로는 생물막 검사가 용이하지 않다(Cramer, 2012).
식품 안전에 대한 관심이 커지면서 생물학적 오염원을 쉽고 빠르게 검사할 수 있는 새로운 검사기술에 대한 요구가 커지고 있다. 다양한 신규 검사기술 중에서 초분광 영상 기술은 신속성과 비파괴적 검사 특성으로 인하여 식품 품질 및 안전 검사를 위한 대안으로서 주목받고 있다(Rahman et al., 2018). 초분광이란 기존 한 지점에 대해 1차원 분광 스펙트럼 정보를 획득하는 분광 기법을 넘어서 2차원 영상 각각의 픽셀에 대해 1차원 분광 스펙트럼을 획득하여 총 3차원 데이터를 얻을 수 있는 기술이다. 초분광 영상 기술은 분광분석을 통하여 물질의 물리화학적 특성을 파악할 수 있을 뿐만 아니라, 영상분석을 통하여 특정 성분의 공간적 분포에 관한 정보를 동시에 알아 낼 수 있기 때문에 눈에 잘 보이지 않는 농산물의 결함이나 오염물질 등을 판별하는데 매우 적합하다(Wu and Sun, 2013; Lim et al., 2016).
Yoon et al. (2009)은 반사광 초분광 영상과 패턴인식 기술을 이용하여 한천 배지 위에 배양한 17종의 균들 중에서 캠필러박터균(Campylobacter)을 99%의 정확도로 판별하였으며, Zhu et al. (2016)은 형광 초분광 영상을 이용하여 옥수수에 오염된 아플라톡신(Aflatoxin) 곰팡이독소가 100 ppb 이상일 때 95.3%의 정확도로 판별하였다. 또한, Peng et al. (2011)은 반사광 초분광 영상 기술을 이용하여 소고기의 부패균 농도를 배양법으로 측정한 값에 매우 근접하게(r2 = 0.95) 예측하였으며, Huang et al. (2013)은 같은 기술을 이용하여 돼지고기의 부패균 농도를 실제 농도와 유사하게(r2 = 0.83) 예측하였다.
본 연구는 농식품 가공시설에서 세균의 교차오염을 발생시키는 주요 원인인 생물막을 초분광 영상 기술을 활용하여 신속하게 검사할 수 있는 가능성을 검토하기 위하여 수행되었다. 초분광 영상 장치는 라인스캔 방식으로 420 - 730 nm 사이의 형광 영상을 획득하여 분석하였고, 생물막은 농식품 가공시설의 주재료로 사용되는 HDPE (high-density polyethylene) 표면에 대장균에 의해 형성된 것을 대상으로 하였다. 또한, 초분광 영상을 이용한 대장균 생물막 검출 정확성을 향상시키기 위하여 기계학습법(machine learning)을 적용하여 초분광 영상을 분석하였다.
Materials and Methods
대장균 생물막 형성
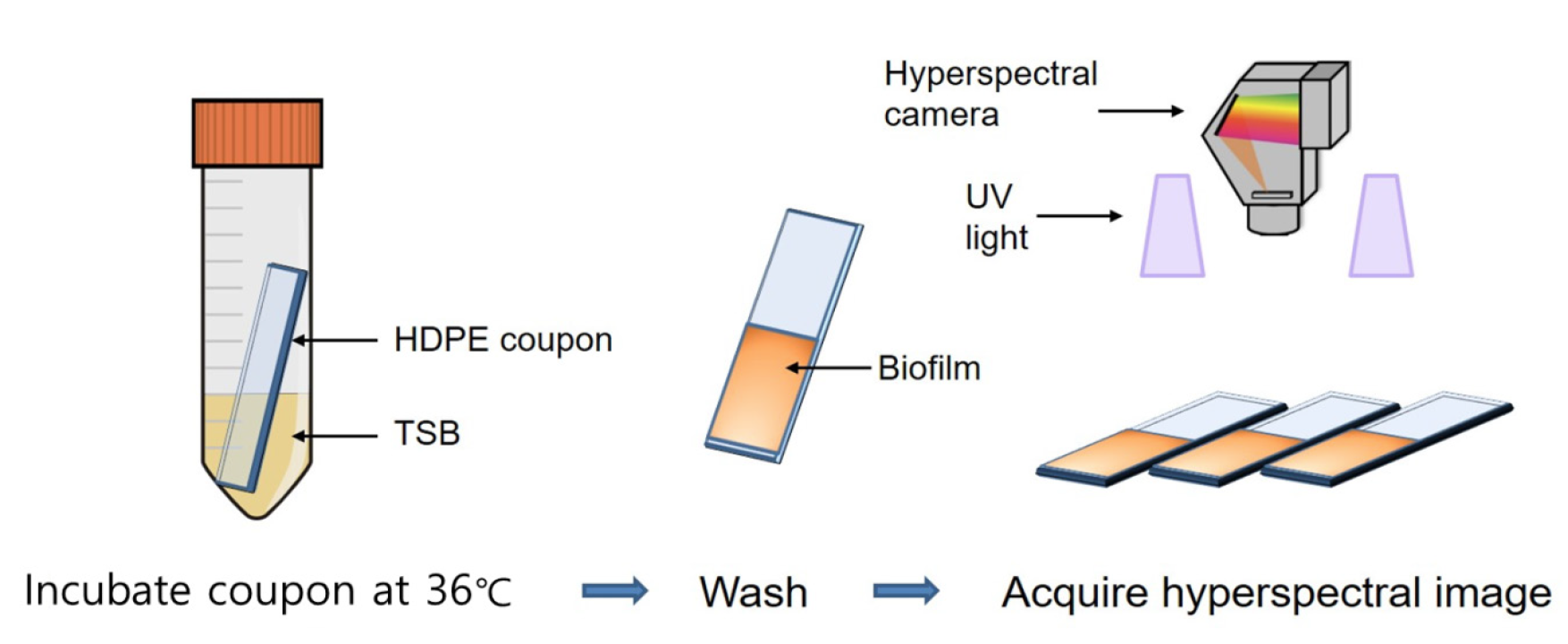
식품 가공 시설에서 많이 사용되는 HDPE 표면 위의 생물막은 한국 미생물 보존센터에서 분양받은 일반 대장균(Escherichia coli [E. coli], KCCM 11234)을 사용하여 형성하였다. 생물막 형성에 사용된 HDPE 조각(20 × 50 × 1 mm3)은 실험 전에 초음파 세척기(WUC-A, DAIHAN Ultrasonic cleaner, Wonju, Korea)를 이용하여 세척하였으며, 실험에 사용된 모든 시약과 배지, 쿠폰은 고압 멸균기(MLS-2420, SANYO, Tokyo, Japan)를 사용하여 121℃에서 15분 간 멸균한 후 사용하였다. 생물막 형성 과정 첫 단계로 분양받은 대장균 종균을 TSB (Tryptic Soy Broth, BD, Franklin Lakes, NJ, USA) 배지에 접종한 다음 36℃에서 24시간 배양하여 활성화 하였다. 대장균 배양액은 다시 백금이로 채취하여 TSA (Tryptic Soy Agar, BD, Franklin Lakes, NJ, USA) 평판배지 표면에 접종하고 36℃에서 24시간 배양하였다. 형성된 단일 집락을 채취하여 PBS 용액으로 현탁액을 제조하였고, 8000 rpm에서 3분간 원심분리(Eppendorf centrifuge 5804 R, Eppendorf, Hamburg, Germany) 한 후 상등액은 피펫으로 제거하고 다시 PBS 용액에 재현탁하는 세척 과정을 총 3번 진행하였다. 균 배양액은 마이크로플레이트 리더기(Infinite M1000, Tecan, Mannedorf, Switzerland)를 사용하여 600 nm에서 흡광도(optical density, OD)를 측정하였고, 배양액 100 μL를 OD값 0.1로 농도 조절한 것을 실험에 사용하였다. HDPE 조각 표면에 대장균 생물막을 형성하기 위해서 TSB 배지 15 mL이 담긴 50 mL 코니칼 튜브에 접종 균수를 102 CFU·mL-1로 조성한 후, 위에서 준비한 HDPE 조각을 코니칼 튜브에 넣고 36℃에서 8일 간 배양하였다(Fig. 1). 24시간 주기로 코니칼 튜브의 배양액은 제거하고 PBS 용액을 넣고 천천히 교반하면서 2회 세척 후 멸균된 TSB 배지 15 mL을 담아 대장균의 생물막 형성에 필요한 영양분을 공급하였다. 대조군으로는 대장균을 접종하지 않은 배지에서 대장균 생물막 형성 과정과 동일하게 처리한 HDPE 조각을 이용하였다.
생물막 부착 균수
생물막 배양을 마친 HDPE 조각은 피펫을 사용하여 3차 증류수로 표면을 세척하면서 부유 세균들을 제거하고, 무균작업대 안에서 조각 표면을 완전히 건조한 후 초분광 영상을 획득하였다. 그 다음, HDPE 조각 표면에 형성된 생물막을 세포 긁개로 회수하여 PBS 용액으로 옮기고 순차희석한 후 표준평판법과 건조필름법을 이용하여 측정하였다. 표준평판법은 표준 한천 배지(Plate Count Agar, Becton Dickinson & Co., Franklin Lakes, NJ, USA)를 이용하였고, 건조필름법은 대장균용 건조 필름(3M Petrifilm E. coli/Coliform Count Plates, 3M, St. Paul, MN, USA)을 이용하였다. 희석액 배양 배지는 36℃에서 24 - 48시간 배양한 후 형성된 집락수를 계산하여 생성된 생물막이 조각 표면에 부착된 정도를 결정하였고 식(1)과 같이 계산하였다.
쿠폰 표면적 당 생성된 대장균수 (log CFU∙cm-2) = (1)
(1)
초분광 영상 시스템
대장균 생물막 분석에 사용된 초분광 영상장치는 고감도의 라인스캔 EMCCD 카메라(MegaLuca, Andor Technology, Belfast, Northern Ireland, UK), 영상 분광장치(VNIR Hyperspec, Headwall Photonics, Fitchburg, MA, USA), C-mount 렌즈(Schneider Optics, Van Nuys, CA, USA), 자체 제작한 365 nm 자외선 LED 광원, 그리고 선형 모터(XSlide, Velmax, Bloomfield, NY, USA)로 구동되는 전동 시료대로 구성된다. 자외선 광원에 의해 시료로부터 발생된 형광은 렌즈를 통과한 다음 분광장치에서 파장별로 나눠지고, 각 파장의 영상은 영상 획득부의 EMCCD 카메라에 의해 분석 가능한 형태로 변환되어 PC에 저장된다. 하나의 초분광 영상은 310 × 502 화소의 공간 해상도를 포함하고 있으며 420 - 730 nm 파장 대역을 균등 분할한 65개의 파장별 영상을 포함한다.
초분광 영상 기술을 이용한 대장균 생물막 검출 가능성을 조사하기 위하여 대장균을 접종한 배지에 담겨있던 시험군 HDPE 조각 60개, 대장균이 없는 배지에서 처리한 대조군 HDPE 조각 30개에서 양면(앞, 뒤)에 대한 초분광 영상 180개를 획득하였다. HDPE 조각의 생물막 초분광 영상은 Fig. 1과 같이 배지 등 다른 물질로 인한 간섭을 줄이기 위하여 PBS 용액으로 2회, 3차 증류수로 1회 세척하고, 무균작업대 내부에서 건조한 다음 획득하였다.
생물막의 형광 초분광 영상은 광원의 불균일성과 초분광 영상 장비의 전기적 잡음 신호 영향을 제거하기 위한 전처리 작업을 거친 다음 분석에 이용하였다. 전처리 작업은 테프론 재질의 기준 백색판(SRT-151, Labsphere, New Hampshire, MA, USA)으로부터 획득한 영상과 카메라 렌즈 덮개를 씌우고 획득한 암흑영상을 각 형광 초분광 영상에 적용하는 Lim et al. (2013)이 사용한 정규화 방법을 이용하였다.
대장균 생물막 검출 방법
전처리 작업을 통해 교정된 초분광 영상으로부터 시험군과 대조군 HDPE 조각의 배지에 담겨있던 영역에서 스펙트럼을 추출하였다. 다음 단계로 추출된 스펙트럼을 단파장을 활용한 t-검정법과, 다파장을 활용한 기계학습법을 통해 판별 분석하였다. 단파장을 이용한 대장균 생물막 검출은 Jun et al. (2010)의 방법과 유사한 과정으로 수행하였다. 대장균 생물막의 스펙트럼과 대조군 스펙트럼 세기의 차가 가장 큰 파장대역을 선정하였다. 선정된 파장대역을 통해 대장균 생물막 영역과 대조군 영역의 밝기 히스토그램을 구한 다음, 두 영역을 나누는 밝기 문턱값을 찾아 전체 영상을 이진화해 대장균 생물막 영역을 검출하였다. 다파장을 활용한 기계학습 방법으로는 의사결정트리(DT, decision Tree), k-최근접 이웃(k-NN, k-nearest neighbor), 선형판별분석(LDA, linear discriminant analysis), 그리고 부분최소제곱 판별분석(PLS-DA, partial least squares-discriminant analysis)방법을 이용하였다. DT, k-NN, LDA, PLS-DA는 판별 분석 기법의 일종으로 DT는 의사결정규칙을 나무구조형태로 도표화한 후 샘플들을 여러 개의 소집단으로 분류하여 클래스를 예측하는 분석 기법이다. k-NN은 특징공간에서 샘플 간의 거리를 기반으로 하여 가장 가까운 거리에 있는 클래스로 샘플을 분류하는 방식이며 LDA는 클래스 간 분산과 클래스 내 분산의 비율을 최대화 하는 방식으로 특징벡터의 차원을 축소하는 기법이다. PLS-DA는 PLSR (partial least square regression)을 기반으로 한 분석 기법으로 문턱값을 이용하여 회귀식으로 예측된 모델을 클래스로 분류한다. 기계학습을 통한 대장균 생물막 예측 모델 개발과 생물막 영역 검출은 공개 통계 소프트웨어인 R (ver. 2019, R Foundation, R foundation, Vienna, Austria)을 이용하여 수행되었다. 생물막 검출 모델 개발을 위해 초분광 영상에서 추출된 스펙트럼 중 절반은 예측 모델을 개발하는데 이용되었고, 나머지 절반 데이터를 이용하여 생물막 예측 정확도를 검증하였다. 예측 정확도는 식(2), 식(3)과 같이 계산하였다.
Control group accuracy (Specificity) = 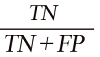 (2)
(2)
Experimental group accuracy (Sensitivity) = 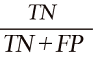 (3)
(3)
TP (true positive)는 생물막이 형성된 영역을 정확하게 예측한 빈도, FP (false positive)는 생물막이 형성되지 않은 영역을 잘못 예측한 빈도, TN (true negative)는 생물막이 형성되지 않은 영역을 정확하게 예측한 빈도, FN (false negative)는 생물막이 형성된 영역을 잘못 예측한 빈도이다. 정확도가 가장 높은 모델을 선정한 후, 모델 개발에 이용되지 않은 초분광 영상에 적용하여 생물막 검출 결과를 확인하였다.
Results and Discussion
대장균 생물막 형성
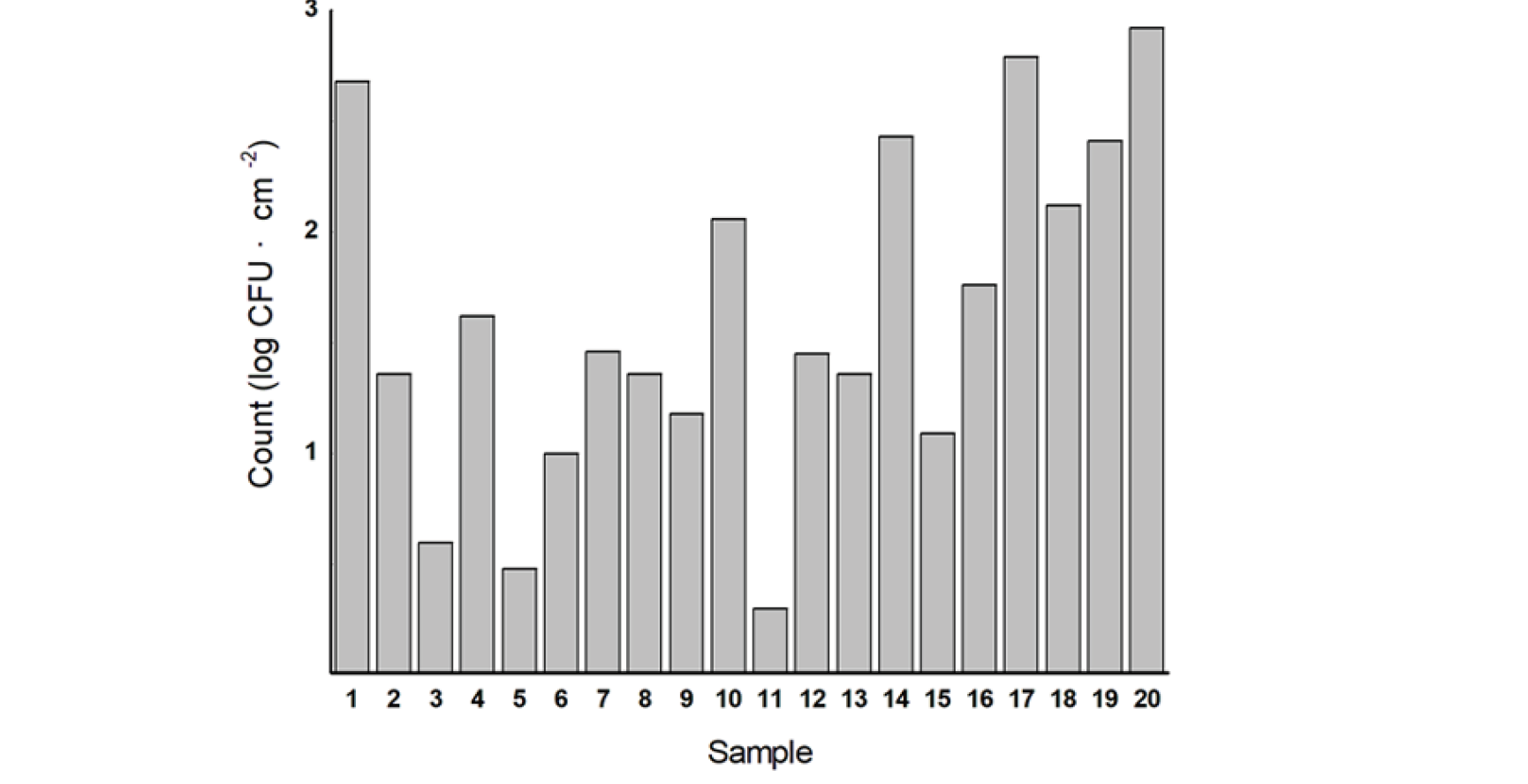
표준평판법과 건조필름법을 이용하여 균 수를 측정한 결과, 시험군 HDPE 조각 120면 중 20면에서 대장균 배양이 성공적으로 이루어졌다. 4면에서는 대장균 배양은 이루어지지 않고 일반 세균 배양만 이루어졌으며, 나머지 면에서는 세균 배양이 이루어지지 않았다. 대장균 배양이 이루어진 20면의 대장균 수는 Fig. 2와 같다. 조각 별 대장균 수는 최소 0.3 log CFU·cm-2에서 최대 2.92 log CFU·cm-2만큼 형성되었다.
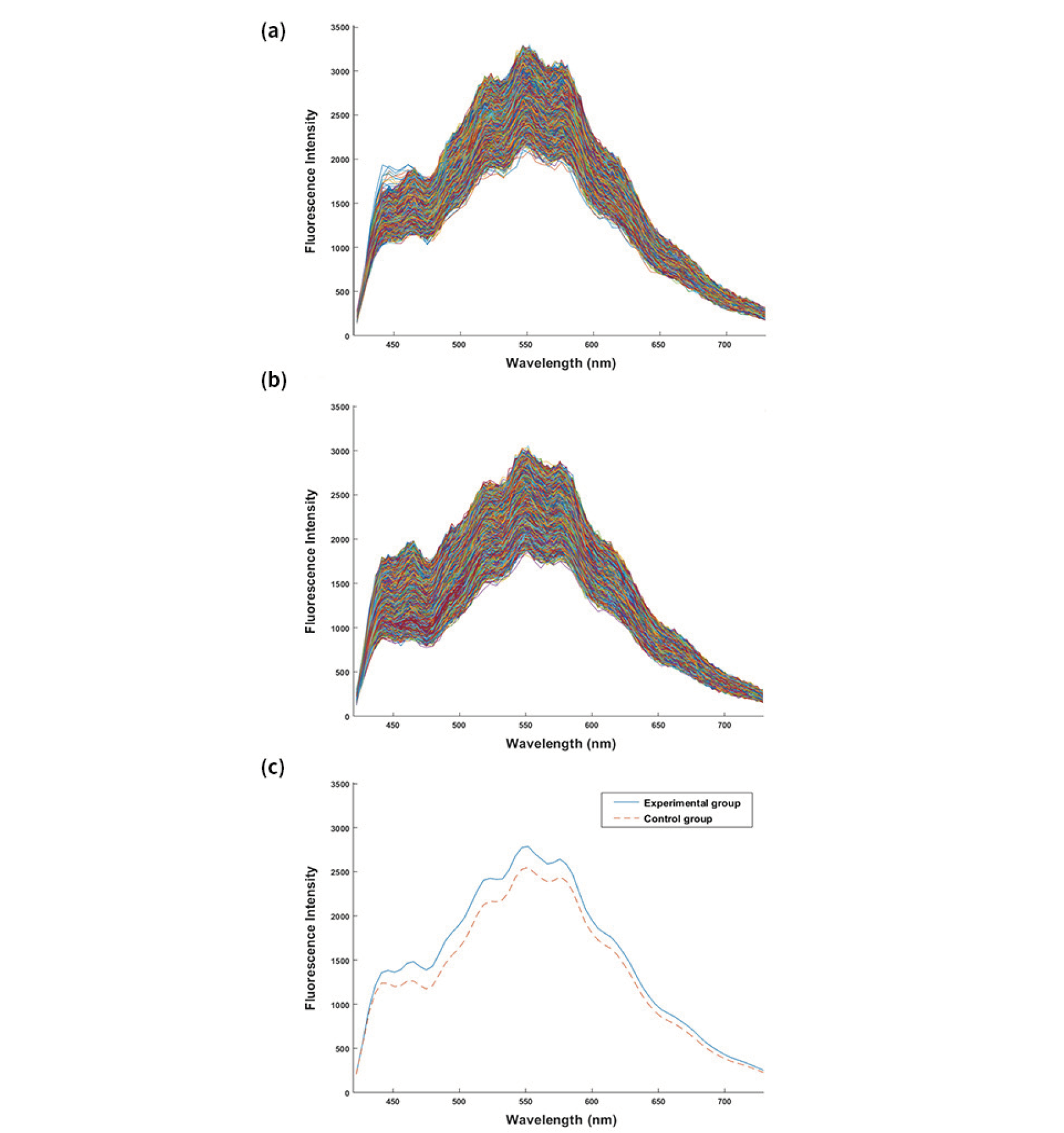
초분광 영상으로부터의 스펙트럼 추출은 배지에 담겨있던 영역인 공기층과 배지의 경계면 아래 부분을 이용하였다. 대장균 생물막 검출 모델 개발을 위해 시험군 중에서도 대장균이 검출되지 않은 면의 영상은 대조군에 포함시켜 분석을 진행하였다. 모델 개발에 사용된 스펙트럼(Fig. 3)은 총 153,720개로, 대장균 배양이 이루어진 HDPE 12면에서 추출된 23,266개의 스펙트럼이 시험군(Fig. 3a)으로 사용되었다. 대조군(Fig. 3b)에는 일반 세균은 검출되었으나 대장균은 검출되지 않은 시험군 HDPE 3면에서 5,586개, 일반 세균과 대장균 모두 검출되지 않은 시험군 HDPE 29면에서 57,352개, 대조군 HDPE 36면에서 추출된 스펙트럼 67,516개로 총 130,454개의 스펙트럼이 사용되었다. Fig. 3의 (c)는 (a), (b)의 평균 스펙트럼으로 측정된 모든 파장 영역에서 대장균 배지에 담겨있던 부위의 형광신호가 대조군 부위보다 높게 나타나는 경향을 확인할 수 있었으나 전체 스펙트럼을 살펴보았을 때 스펙트럼 별 편차가 심한 것으로 보아 일부 파장만으로 두 그룹을 판별하는 것은 어려울 것으로 예측되었다.
대장균 생물막 예측 모델 개발
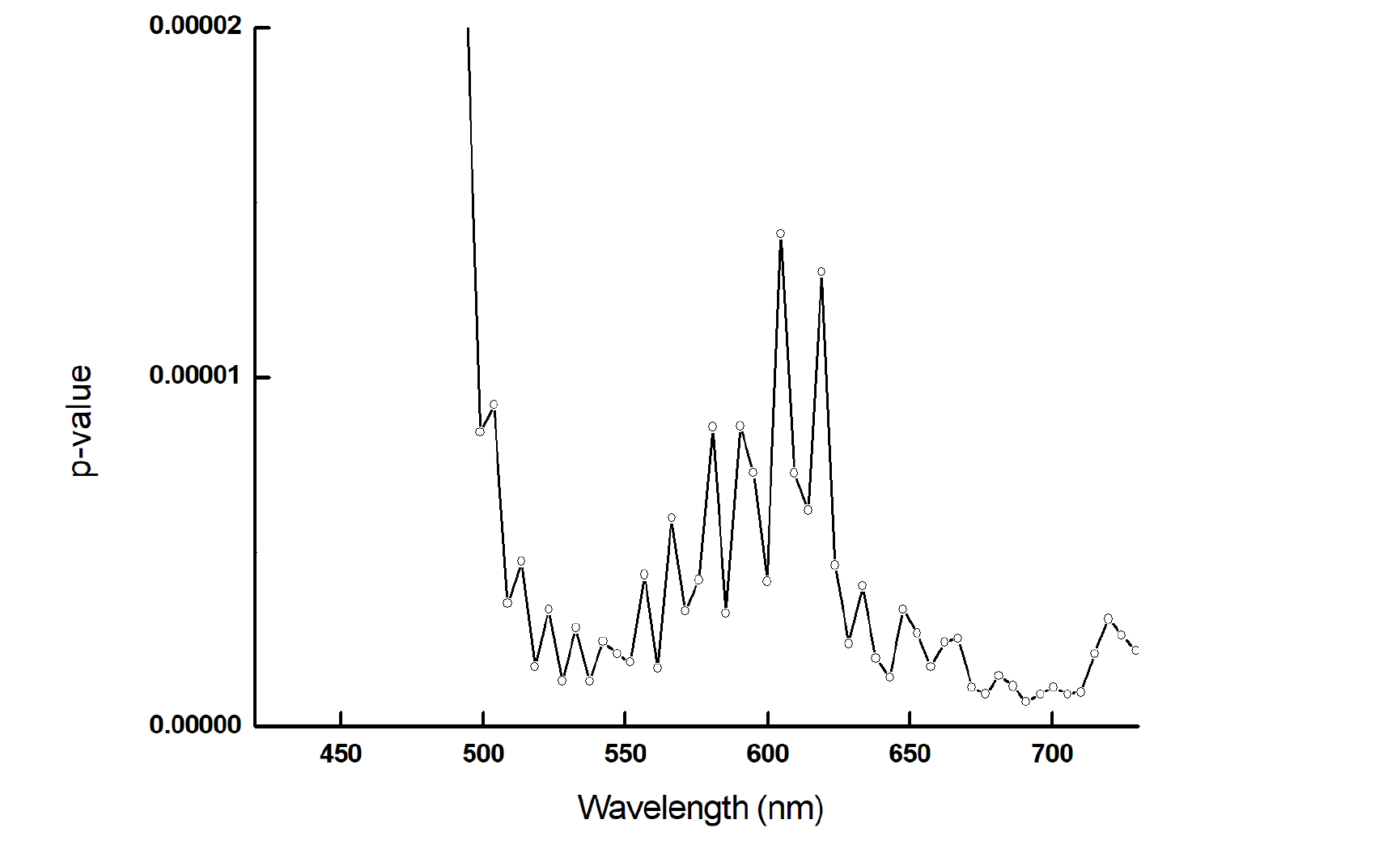
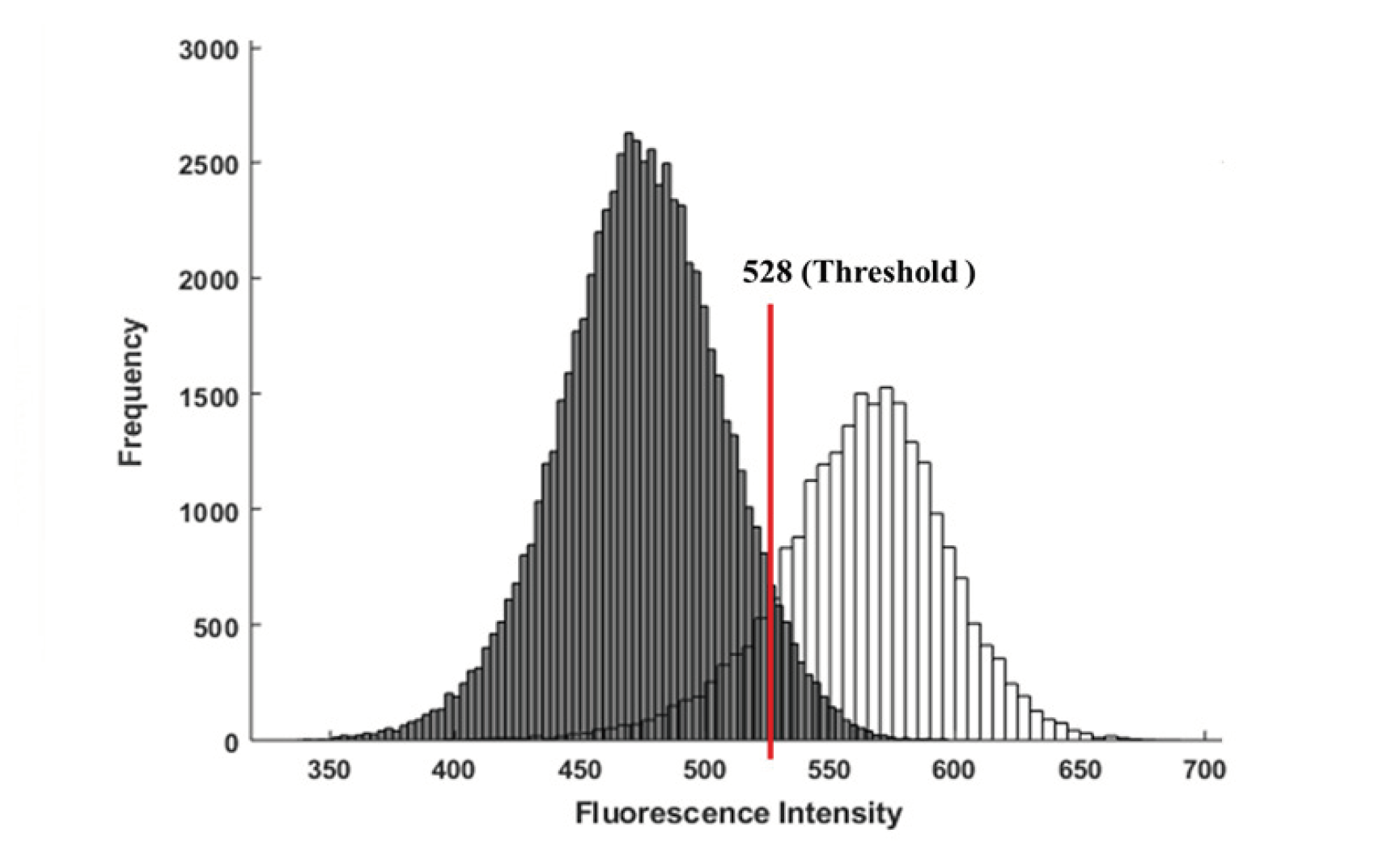
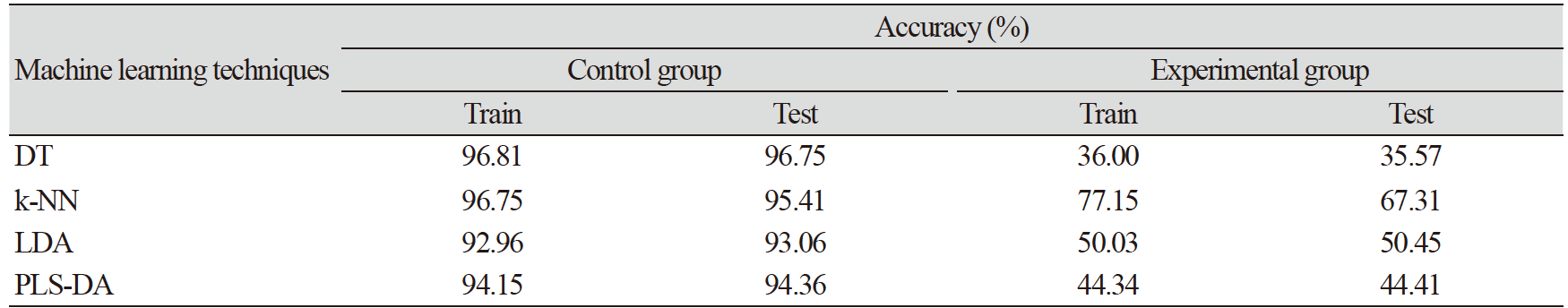
대장균 생물막 예측 모델은 단파장 영상을 이용한 t-검정법과 다파장 영상을 이용한 기계학습 방법을 적용하여 개발하였다. 먼저, 단파장 영상을 이용한 대장균 생물막 검출은 대장균 생물막의 스펙트럼과 대조군 스펙트럼에서 가장 큰 차이를 보이는 파장을 t-검정법을 이용하여 구하였다. 두 영역의 스펙트럼에서 가장 큰 차이를 나타내는 파장은 690 nm로 조사되었다(Fig. 4). 두 영역의 690 nm 형광 영상에 대해 동일 밝기 값을 갖는 화소의 빈도수를 나타내는 히스토그램은 Fig. 5와 같다. 그림에서 두 영역을 구분하는 밝기 값은 528이었으며, 이 값을 문턱값으로 적용하여 판별 분석한 결과, 생물막 예측 정확도는 대조군에서 94.53%, 시험군에서 29.54%의 정확도를 나타냈다. 다파장을 이용한 기계학습 방법의 경우, 판별 분석에 많이 사용되는 DT, k-NN, LDA, PLS-DA 방법을 사용하였다. 시험군의 스펙트럼은 1로, 대조군 스펙트럼은 0으로 클래스를 나눈 후 판별 분석한 결과는 Table 1과 같다. 검정(Test) 그룹에서의 시험군에 대한 LDA 분석의 정확도가 50.03%, PLS-DA 분석의 정확도가 44.34%, DT 분석의 정확도가 36%로 다파장을 이용하였을 경우 단파장을 이용한 t-검정법보다 높은 정확도를 나타내었다. 특히, k-NN 분석을 했을 경우 학습(Train) 그룹의 분류 정확도는 대조군에서 96.75%, 시험군에서 77.15%의 정확도를, 검정(test) 그룹에서는 대조군에서 95.41%, 시험군에서 67.31%의 정확도를 나타내었다. 모든 모델에 대해서 대조군의 정확도가 시험군보다 높음을 확인할 수 있는데 이는 시험군 데이터 수가 대조군에 비해 적은 것에서 기인한 것으로 판단된다. 대장균 검출 모델 개발에는 t-검정법보다 기계학습 방법이 더 효과적임을 확인하였고 그 중에서도 k-NN 분석법이 가장 높은 성능을 나타냈다. Fig. 3에서 알 수 있듯이 같은 군 내에도 스펙트럼 별 편차가 크다 보니 임계 값을 통해 분류하는 DT 분석법이나 선형모형인 LDA와 PLS-DA 분석법의 성능이 낮게 나타난 것으로 판단되며 생물막의 경우, 미생물 군락에 의해 소공간에서 생겨나 점점 확대되어가는 구조를 가지기 때문에 샘플간의 거리를 기반으로 분류하는 방식인 k-NN 분석법이 적합한 것으로 판단된다.
대장균 생물막 검출
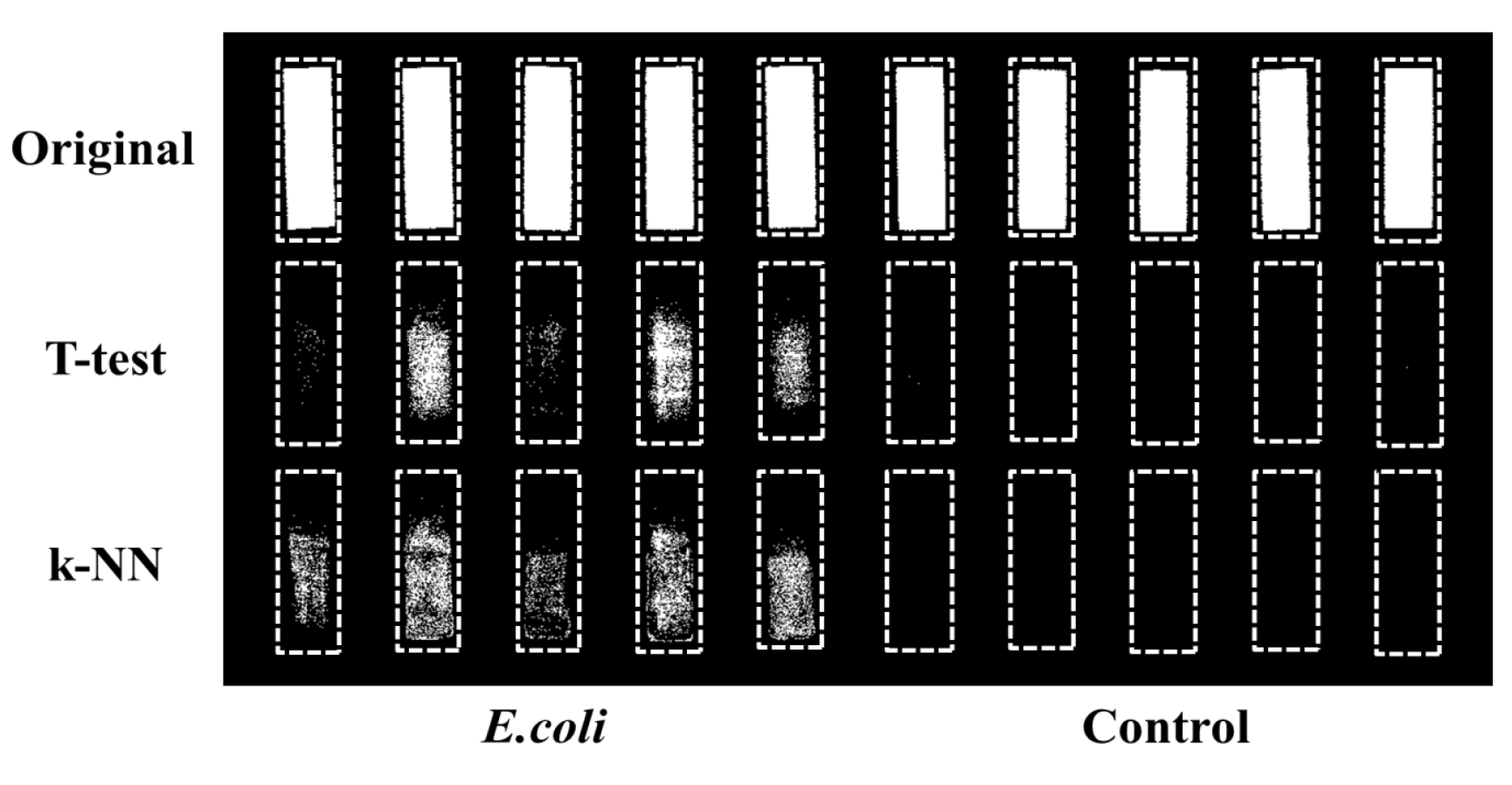
위에서 얻어진 단파장 모델과 기계학습 모델을 초분광 영상을 구성하는 모든 화소에 적용하여 생물막 여부를 예측하였다. 영상에서 ROI (region of interests) 영역을 설정하여 쿠폰 영역과 배경 영역을 이진화하고, 쿠폰 영역에 모델을 적용하여 대장균이 검출된 영역과 검출되지 않은 영역에 대한 이진화 영상을 Fig. 6과 같이 획득하였다. Fig. 6의 쿠폰들은 모델 개발에 사용되지 않은 단파장을 이용한 분석 결과와 k-NN 기계학습 방법을 이용한 분석 결과가 나타나 있다. 두 분석방법 모두 육안으로 판별이 어려웠던 배지에 담겨있던 부위의 대장균 생물막을 성공적으로 검출하였으며, 기계학습을 적용했을 경우에 경계면 아래 부분을 좀더 정확히 검출하는 것을 확인할 수 있었다.
초분광 영상을 이용한 기존의 미생물 또는 생물막 검출 결과를 보면, Jun et al. (2009)은 단파장을 이용한 스테인리스 금속막 표면의 대장균 생물막을 검출하였는데, 이때 생물막의 대장균 수는 7 log CFU·cm-2 이상인 것으로 보고되었다. 또한, 같은 연구 그룹이 HDPE 조각 표면의 대장균 생물막을 초분광 영상에서 단파장 또는 2개 파장의 비를 이용하여 검출하였는데, 이때 생물막에 포함된 대장균 수는 7.56 log CFU·cm-2인 것으로 보고되었다. 본 논문에서는 단파장과 다파장을 이용하여 검출하였으며 다파장을 활용한 기계학습법의 경우, 대장균 생물막 최소 검출 한계는 0.3 log CFU·cm-2으로 기존 연구결과와 비교하였을 때 우수한 것으로 나타났다. 기계학습법의 경우, 기존 분석 방법에 비해 모델 개발에 사용되는 데이터 용량이 크고 모델 학습 시간이 오래 걸리는 단점이 있으나 모델 개발에 필수적인 일부 파장대역만을 선정해 학습에 필요한 메모리 사용량을 줄이는 최적화 기법을 통해 이러한 문제점을 해결할 수 있으리라 판단된다. 또한, 오검출률을 줄이기 위해서는 추가 실험을 통해 더 많은 샘플을 확보하여 모델 학습을 진행하는 등 추후 연구가 필요한 것으로 보여진다.
Conclusion
본 연구는 농식품 가공시설에서 세균의 교차오염을 일으키는 주요 원인인 생물막을 초분광 영상 기술 및 기계학습 분석 기법을 활용하여 신속하게 검사할 수 있는지를 구명하기 위하여 수행되었다. 초분광 영상은 라인스캔 방식의 장치를 이용하여 420 - 730 nm 사이의 형광 영상을 획득하여 분석하였고, 생물막은 농식품 가공시설의 주재료로 사용되는 HDPE 표면에 대장균에 의해 형성된 것을 대상으로 하였다. 이러한 연구결과를 요약하면 다음과 같다.
첫째, 초분광 영상을 k-NN 기계학습 기법으로 분석하였을 때 90% 이상의 높은 정확도로 대장균 생물막 영역을 예측할 수 있었다.
둘째, 검출된 대장균 생물막 최소 검출 한계는 0.3 log CFU·cm-2으로 기존 연구결과 보다 우수한 것으로 조사되었다.
셋째, 기계학습에 의한 대장균 생물막 예측 모델을 영상의 전체 화소에 적용하였을 때 생물막 영역을 정확히 검출할 수 있는 것으로 나타나 초분광 영상을 이용한 현장에서의 실시간 생물막 검출 가능성을 확인할 수 있었다.
Authors Information
Ahyeong Lee, https://orcid.org/0000-0002-8524-6282
Youngwook Seo, https://orcid.org/0000-0003-2140-5333
Jongguk Lim, https://orcid.org/0000-0002-2501-4367
Saetbyeol Park, https://orcid.org/0000-0001-5931-2699
Jinyoung Yoo, https://orcid.org/0000-0002-6370-7973
Balgeum Kim, https://orcid.org/0000-0003-4946-9152
Giyoung Kim, https://orcid.org/0000-0002-5186-3223