Introduction
논 생태계는 일정한 시기(봄 - 여름)에 담수되는 인공적인 내륙습지이며 하천 및 저수지와 연결되어 있어서 다양한 수서생물종이 산란 및 서식하는 습지생태계의 독특한 형태이다(Saito, 1988; Kim et al., 2016). 지구상 전체 습지의 약 18%를 차지하고 있는 논 생태계는 람사르(Ramsar) 협약에 의해 습지생태계 중의 하나로 인식되면서 생물다양성 보전 측면에서 많은 주목을 받고 있다(Bang et al., 2010; Ko et al., 2016; Oh et al., 2019). 최근 세계적으로 환경오염 및 생물다양성의 보전 및 증진을 위해 습지를 활용한 효율적인 이용방안이 대두되고 있으며 ʻ습지 시스템으로서 논의 생물다양성 증진 결의문(Enhancing Biodiversity in Rice Paddies as Wetland System)’이 람사르 회의에서 채택되면서 논을 활용한 생물다양성 증진에 대한 관심이 높아지고 있다(Ramsar-Scientific & Technical Review Panel, 2008).
국내에서 논 생태계의 생물조사는 1960년대부터 논의 모기 발생량을 확인하기 위한 보건위생연구소의 연구조사가 처음이다(Kang et al., 2013). 1994년 부터 농촌진흥청 농업과학기술원에 신설된 농업생태과에서 본격적인 논 수서생물 연구조사가 수행되었으며, 2008년 창원 람사르 총회 이후, 전문가 영역을 넘어서 민간 단체에서 논습지 네트워크를 구성하고 논 생물 조사를 수행하고 있다(Jang et al., 2010; Kang et al., 2013; Sagong et al., 2014). 현재도 논 생태계 실태조사를 통해 논 환경에 서식하는 양서·파충류와 거미류 및 수서곤충의 출현을 조사하고 있으며 이를 통해 기후변화에 따른 생물군의 출현 일시 및 출현 지역 변화를 파악하고 있다(Eea et al., 2019; Kim, 2021).
이러한 논 생태계 생물의 실태조사는 기본적으로 개체를 직접 채집하여 관찰하는 전통적인 방식을 사용하고 있다. 수서곤충의 경우에는 수중트랩 및 채반을 이용하여 개체를 채집하고 있으며, 양서류의 경우에는 조사자가 직접 현장에서 개체를 촬영하거나 개체의 울음소리를 듣고 출현을 판단하고 있다(Kang et al., 2020). 하지만 이러한 전통적인 채집방법들은 매우 노동집약적이며, 조사시기에 따른 차이가 크게 나타난다. 수서곤충 채집의 경우에는 잠자리 및 하루살이와 같이 유생에서 성충으로 우화하여 논 수생태계를 떠날 경우에는 해당 개체의 존재여부를 파악하기 매우 어렵다. 또한 유생을 채집하기 위한 수중트랩은 수서곤충 유인을 위한 어분이 수서곤충 뿐만 아니라 주변의 양서류 유생(올챙이)까지 유인하기 때문에 채집효율을 감소시키는 원인으로 작용할 수 있다. 양서류의 경우에는 매우 넓은 범위에서 개체를 탐색해야 하기 때문에 조사수행에 매우 많은 시간이 소요될 수 있으며 개체수가 많지 않은 경우에는 논 생태계에 존재하는 개체를 탐색하기 매우 어렵다.
환경유전자를 이용한 생물탐색법은 이러한 전통적인 생물탐색법의 문제점과 어려움을 해소하고자 제시되었다(Ficetola et al., 2008; Yoccoz, 2012; Taberlet et al., 2018). 환경유전자(environmental DNA, eDNA)는 물, 공기, 토양 등 다양한 환경에서 대량으로 존재하는 유전물질을 의미하며 이러한 유전물질에는 해당 환경에 서식하는 생물들의 흔적들이 남아있기 때문에 직접 채집하지 않고 생물의 존재 여부를 판단하는 단서로써 활용될 수 있다(Jerde et al., 2011; Takahara et al., 2013; Bohmann et al., 2014). 현재 환경유전자를 이용한 생물탐색은 어류 분류군에서 가장 활발하게 사용되고 있으며, 국내에서도 멸종위기종(흰수마자)을 탐색하는데 하천에 분포하는 환경유전자를 활용하였다(Takahara et al., 2013; Edmunds et al., 2019; Kim et al., 2020).
본 연구에서는 논 수생태계에 존재하는 환경유전자를 기반으로 해당 수생태계에 서식하는 수서곤충과 개구리 군집을 파악하고 이를 검증하여 향후 논 수생태계 생물실태 조사의 적용 가능성을 판단하였다.
Materials and Methods
Survey site and eDNA sampling
대상 조사지역은 충남 논산시 연산면 송정리에 위치하는 논을 대상으로 하였으며, 사람의 접근과 외부로부터 환경유전자 유입이 가장 적을 뿐만 아니라 산과 인접하여 양서류의 산란이 용이한 논을 선정하였다(Fig. 1). 양서류의 유생이 성체가 되는 시기를 고려하여 7월 중에 1주일 동안 비가 오지 않은 시기에 eDNA를 채집하였으며 eDNA는 Sterivex filter (1 μm pore size, Milipore, Massachusetts, USA)와 50 mL 주사기를 사용하여 현장에서 즉시 여과 및 채집하였다. 논의 형태에 따라서 최소 2곳 이상에서 논 내부의 물에 존재하는 eDNA를 채집하였으며 논 인근에 위치하며 농업용수로 사용하고 있는 하천의 eDNA를 논으로 유입되기 전 위치에서 동일한 방법으로 채집하였다. 채집 즉시 Sterivex filter에 RNAlater (Thermo Fisher, Massachusetts, USA)를 1 mL주입하고 주입구와 배출구를 Parafilm으로 밀봉하였다. eDNA를 채집한 Sterivex filter는 저온(4℃)상태를 유지하며 실험실로 운반하였으며, 실험실에서 24시간 이내에 eDNA를 추출하였다.
eDNA extraction and PCR amplification
실험실로 운반된 Sterivex filter로부터 eDNA를 추출하였으며, eDNA 추출방법은 일본의 eDNA 학회(eDNA society)에서 제시한 추출법에 따라서 수행하였다(Minamoto et al., 2021). eDNA를 추출한 과정은 다음과 같다. 시료당 380 μL의 세포용해 용액(Buffer ATL)과 20 μL의 단백질 분해 효소(Protease K)를 혼합하여 Sterivex filter 내부에 주입하고 56℃로 설정된 항온기에서 30분 동안 반응시켰다. 반응이 끝난 Sterivex filter를 3,000 G 속도로 2분 동안 원심분리하여 추출된 eDNA 원액을 회수하였다. 회수된 eDNA 원액에 세포용해 용액(Buffer AL)을 첨가하고 56℃에서 10분 동안 추가 반응하였다. 반응이 끝난 eDNA 원액을 Column tube에 650 μL를 옮겨 담고 원심분리하여 column과 eDNA를 결합하였다. Column에 결합되어 있는 eDNA를 세척용액(Buffer AW1, AW2)으로 세척한 후 100 - 200 μL으로 용출하여 1.5 mL micro-tube에 보관하였다. 추출이 완료된 eDNA 시료는 분석하기 전까지 초저온 냉동고(Deep freezer, -80℃)에서 보관하였다.
논 수생태계 eDNA에 존재하는 양서류와 저서성 대형무척추동물의 유전자를 확인하기 위해 Sakata 등(2021) 가 제시한 primer를 이용하여 mitochondrial 16S rDNA (mt16S rDNA) 유전자를 증폭하였다. 유전자 증폭 과정은 다음과 같다. DNA 중합효소는 2X GainBlueTM Hot Start Max Master Mix PCR Master Mix (Gainbio, Daejeon, Korea)를 사용하였으며 primer는 forward와 reverse를 10 pmol·μL-1의 농도로 만들어 첨가하였다. 주형(template) eDNA는 3 μL를 첨가하였으며 PCR Thermal Cycler (LongGen, Hangzhou, China)를 사용하여 PCR을 수행하였다. PCR 증폭 조건은 95℃에서 3분간 pre-denaturation을 시행한 후 95℃에서 20초 간 denaturation, 58℃에서 20초 간 annealing, 72℃에서 1분 간 extension을 40 cycle반복한 뒤, 마지막으로 72℃에서 5분간 final extension 하였다.
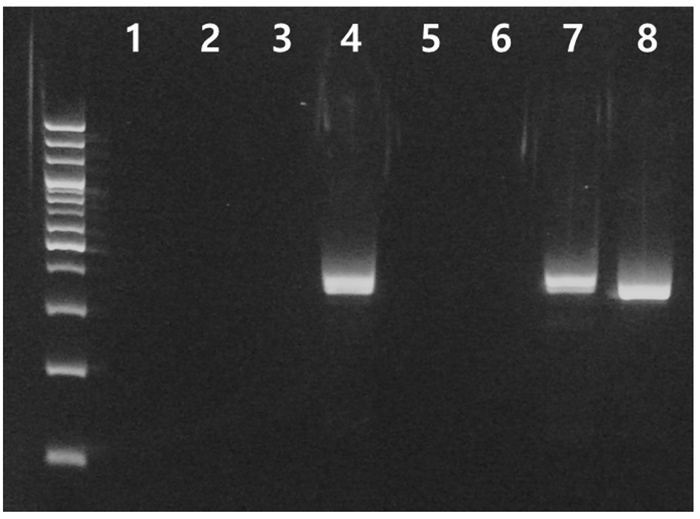
PCR 증폭 산물은 E-gelTM Power Snap Electrophoresis system (Thermo Fisher Scientific, Massachusetts, USA)을 사용하여 2% agarose gel에서 전기영동을 시행한 후 E-gelTM Power Snap Camera (Thermo Fisher Scientific, Massachusetts, USA)로 증폭산물을 확인하였다.
eDNA meta-barcoding analysis
Meta-barcoding 분석은 Illumina miseq 분석 system을 사용하였으며 mitochondrial 16S rDNA (mt16S rDNA) primer를 기반으로 Illumina miseq adapter 염기서열을 부착하여 개구리 및 수서곤충의 mt16S rDNA 유전자를 1차 PCR 증폭하였다. 1차 PCR 증폭은 다음의 과정을 통해 진행되었다. PCR 증폭 조건은 95℃에서 3분간 pre-denaturation을 시행한 후 95℃에서 20초 간 denaturation, 58℃에서 20초 간 annealing, 72℃에서 1분 간 extension을 40 cycle반복한 뒤, 마지막으로 72℃에서 5분간 final extension을 시행하였다.
증폭산물은 E-gelTM Power Snap Electrophoresis system (Thermo Fisher Scientific, Massachusetts, USA)을 사용하여 2% size selection gel에서 전기영동을 시행한 후 대상 증폭 산물을 gel에서 추출하였다. 1차 증폭된 PCR 증폭산물의 농도가 meta-barcoding분석을 하기에 충분한지 quality control (QC)을 진행하였으며 QC를 통과하지 못한 시료는 1차 PCR을 추가 진행하여 증폭산물의 농도를 조절하였다. QC를 통과한 1차 PCR 증폭산물은 Illumina platform 기반의 paired end 방법으로 염기서열을 분석하였다. Illumina platform에서 분석된 염기서열은 실시간분석(real time analysis, RTA) 통합 프로그램을 통해 platform의 제어와 염기분배(base calling)를 진행하였다. 이진법으로 분석된 염기분배 결과는 Illumina 소프트웨어 패키지 bcl21fastq를 사용하여 FASTQ 형태로 변환하였다. FASTQ 파일의 metabarcoding 초기분석자료(rawdata)는 Mac iOS 기반의 Terminal (Version 2.11 (440), Apple Inc, California, USA)의 명령어를 입력하여 FASTA 파일로 변환 후 miseq adapter를 모두 제거하였다.
Meta-barcoding 결과를 검증하기 위해 일부 분류군에 대하여 mt 16S rDNA 유전자의 계통분석을 수행하였다. NCBI의 GenBank에서 개구리목 및 절지동물의 mt 16S rDNA 유전자 정보를 내려 받았으며 MEGA (version 11, Institute of Molecular Evolutionary Genetics, Pennsylvania, USA)를 이용하여 유전자를 정렬(alignment) 하였다. 정렬된 유전자는 Maximum likelihood 방법과 Tamura-Nei 모델을 사용하여 계통수를 분석하였다(Tamura and Nei, 1993; Tamura et al., 2021).
Field survey for frog and aquatic insect
eDNA를 채집한 지역에서 출현하는 개구리 및 수서곤충 개체를 채집하여 eDNA 기반으로 발견된 개체들을 검증하였다.논 환경에 서식하는 개구리 개체를 확인하기 위해 “농업분야 생태환경 조사평가 기준 및 매뉴얼”의 양서류·파충류 조사방법에 따라 조사를 수행하였다(Kang et al., 2020). 낮 시간대에 대상지역의 논 내부와 논두렁, 논수로에서 개구리 성체를 육안으로 조사하였으며 발견된 개체를 사진으로 촬영하여 종을 확인하였다(RDA, 2019a).
논 서식 수서곤충 개체는 대상지 논에 5개 수서트랩을 설치하였다(Yoon et al., 2017). 수서트랩은 일회용 플라스틱 컵에 떡밥(Ilsungsa, Sejeong, Korea) 3 g과 케미라이트(Mistrall, Hangzhou, China)를 유인제로 사용하여 24시간 동안 설치하였다(Fig. 1B). 회수한 개체는 실험실로 운반하여 80% 알콜에 보관하였으며 실체현미경을 이용하여 형태적 특징을 관찰하고, 도감(RDA, 2019b)에 따라 종(species)수준까지 동정하였다. 수서곤충의 경우에는 포충망을 이용하여 비행하는 성체를 함께 채집하여 종을 동정하였다.
Results and Discussion
eDNA metabarcoding analysis
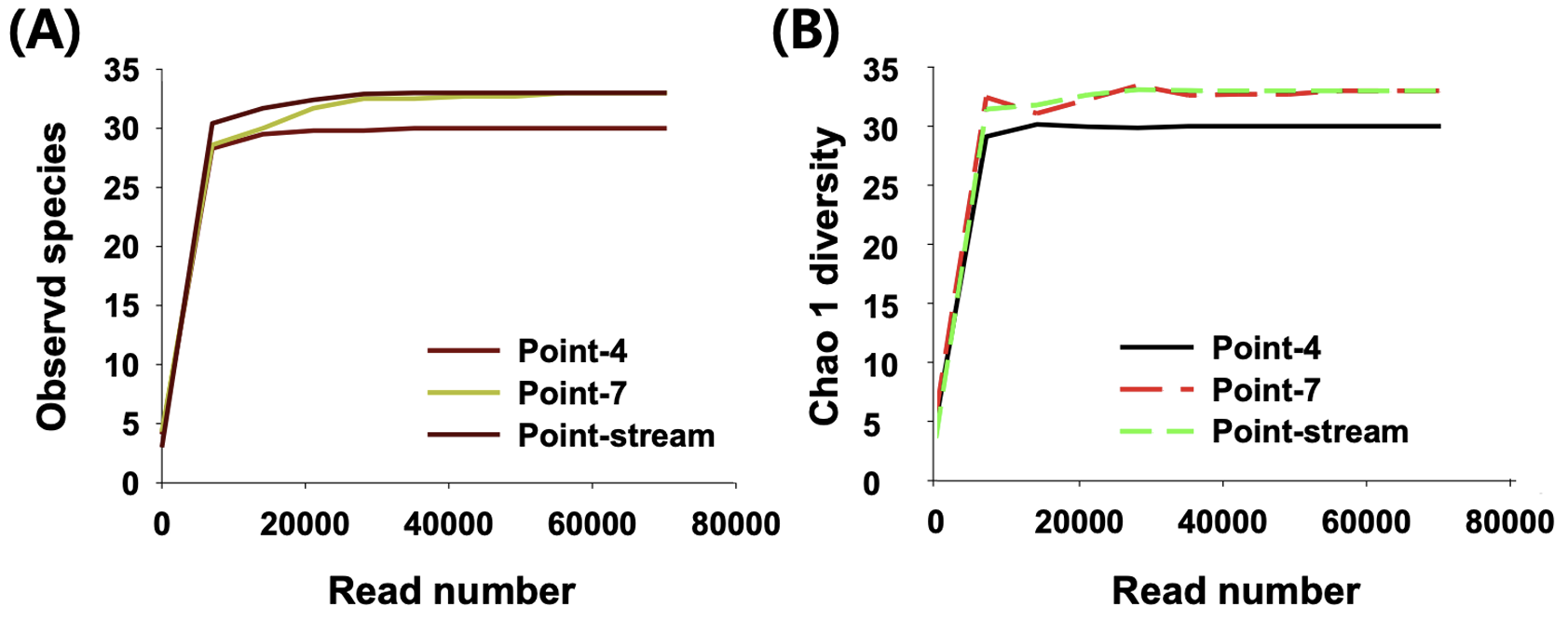
논 환경에서 채집된 eDNA에서 개구리와 수서곤충의 mitochondrial 16S rDNA (mt 16S rDNA)를 증폭한 결과 논 내부 7개 point 중에서 2개 point 에서만 유전자가 증폭되었으며 대상 논으로 공급되는 하천수에서도 mt 16S rDNA 유전자가 증폭되었다(Fig. 2). Metabarcoding을 통해 군집을 분석한 결과 약 35,000 read 이후부터 새로운 분류군이 발견되지 않았으며, 총 30 - 33개의 서로 다른 염기서열이 발견되었다(Fig. 3). Chao 1 diversity는 30 - 33, Shannon diversity는 1.9 - 2.5의 범위를 나타냈으며 point-7에서 가장 많은 종의 유전자가 발견되었다. 하지만 3개 point에서 발견된 유전자의 수와 다양도 지수는 큰 차이를 나타내지 않았는데, 이는 하나의 논에서 발견되었을 뿐만 아니라 논수로가 논과 매우 인접하여 존재하기 때문에 발견되는 분류군의 유전자가 유사한 것으로 판단된다.
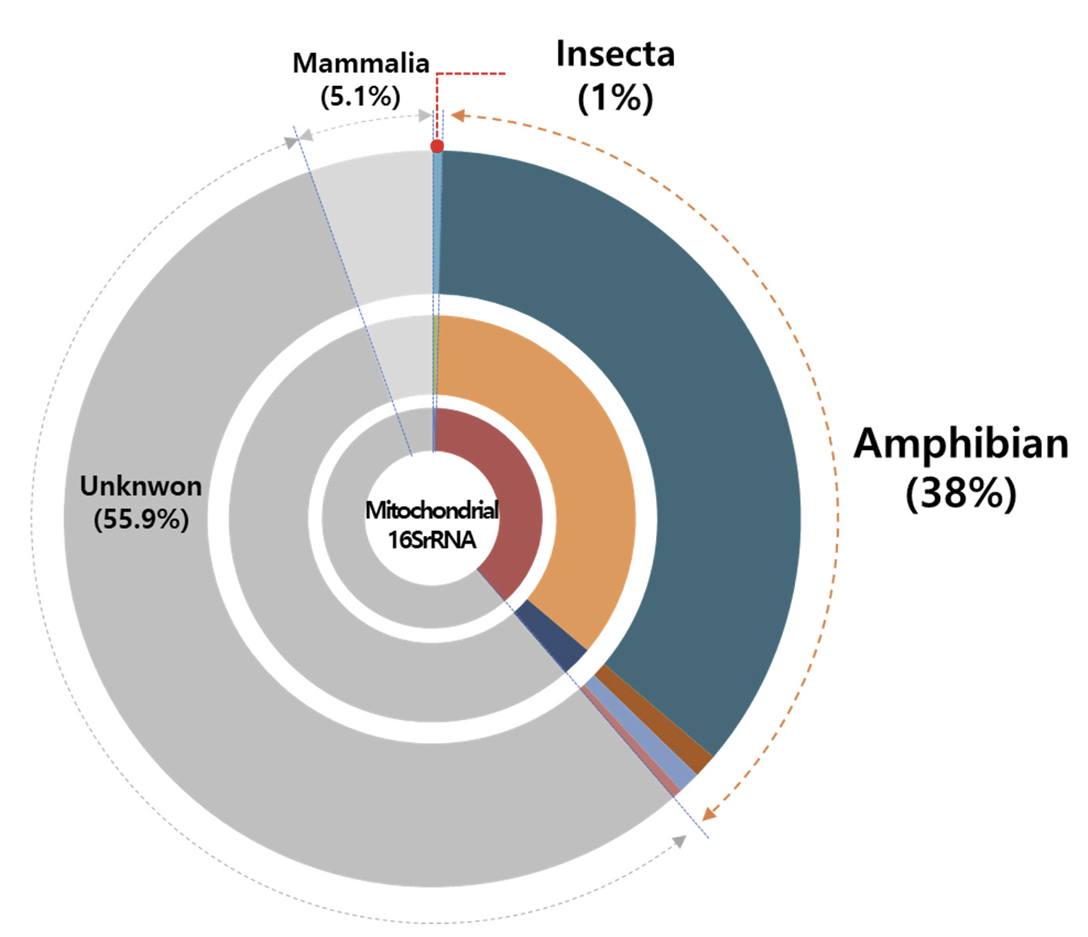
3개 지점에서 발견된 mt 16S rDNA 유전자에서 개구리 유전자는 38%로 생물 분류군 중에서 가장 많은 비율을 차지하였으며, 포유류가 5.1%, 곤충이 1.0%로 가장 낮은 비율을 차지하였다(Fig. 4). 이와 함께 BLAST 분석에서 분류군을 확인할 수 없는 Unknown 염기서열이 전체의 55.9%로 많은 비율을 차지하였다. 개구리는 논과 인접하여 서식하는 생물 중에서 생물크기가 가장 크고 활동범위가 매우 넓다. 이로 인해 논 환경에 존재하는 유전자의 양 또한 많기 때문에 정수환경에서 유전자가 쉽게 발견될 수 있다(Takeuchi et al., 2019). 반면에 수서곤충은 개구리 보다 생물크기가 월등히 작고, 활동성 또한 개구리 보다 높지 않아서 상대적으로 매우 적은 량의 유전자가 존재하게 된다(Herder et al., 2014). 포유류는 개구리 보다 생물크기가 크고 발생하는 상피세포의 양이 더욱 많지만 논 보다는 밭과 같은 경작지에서 주로 서식하며, 논 환경에 직접적으로 연결되는 경우가 적기 때문에 포유류가 직접적으로 유전자를 논 환경에 떨어뜨리는 경우는 드물다(Lee et al., 2017). 이러한 낮은 비율의 포유류 유전자는 하천수가 논 수로를 통해 논으로 유입되는 과정에서 하천수에 녹아 있는 포유류의 환경유전자가 논으로 유입된 것으로 판단된다.
Unknown 염기서열은 megablast (highly similar sequences) 최적화에서는 일치하는 유전자가 없는 것으로 나타나지만, blastn (somewhat similar sequences)으로 최적화하여 BLAST 분석을 수행하였을 때 Homo sapiens (사람)의 유전자와 가장 유사하였다. 이는 논의 특성상 사람이 진입하여 모내기 및 농업활동을 수행하며, 이 과정에서 사람의 상피세포가 논으로 유입될 가능성이 존재한다. 하지만 사람의 상피세포가 논 환경에 노출되었을 때, 빛(자외선)과 온도, 그리고 박테리아의 분해활동으로 인해 유전자 염기서열이 지속적으로 분해된다(Hebsgaard et al., 2005; Shapiro, 2008; Zulkefli et al., 2019). 이로 인해 염기서열이 부분적으로 손실되거나 단편화(fragmentation) 되어 정확한 동정(identification)이 어려울 수 있다(Lindahl, 1993).
Community analysis based on eDNA
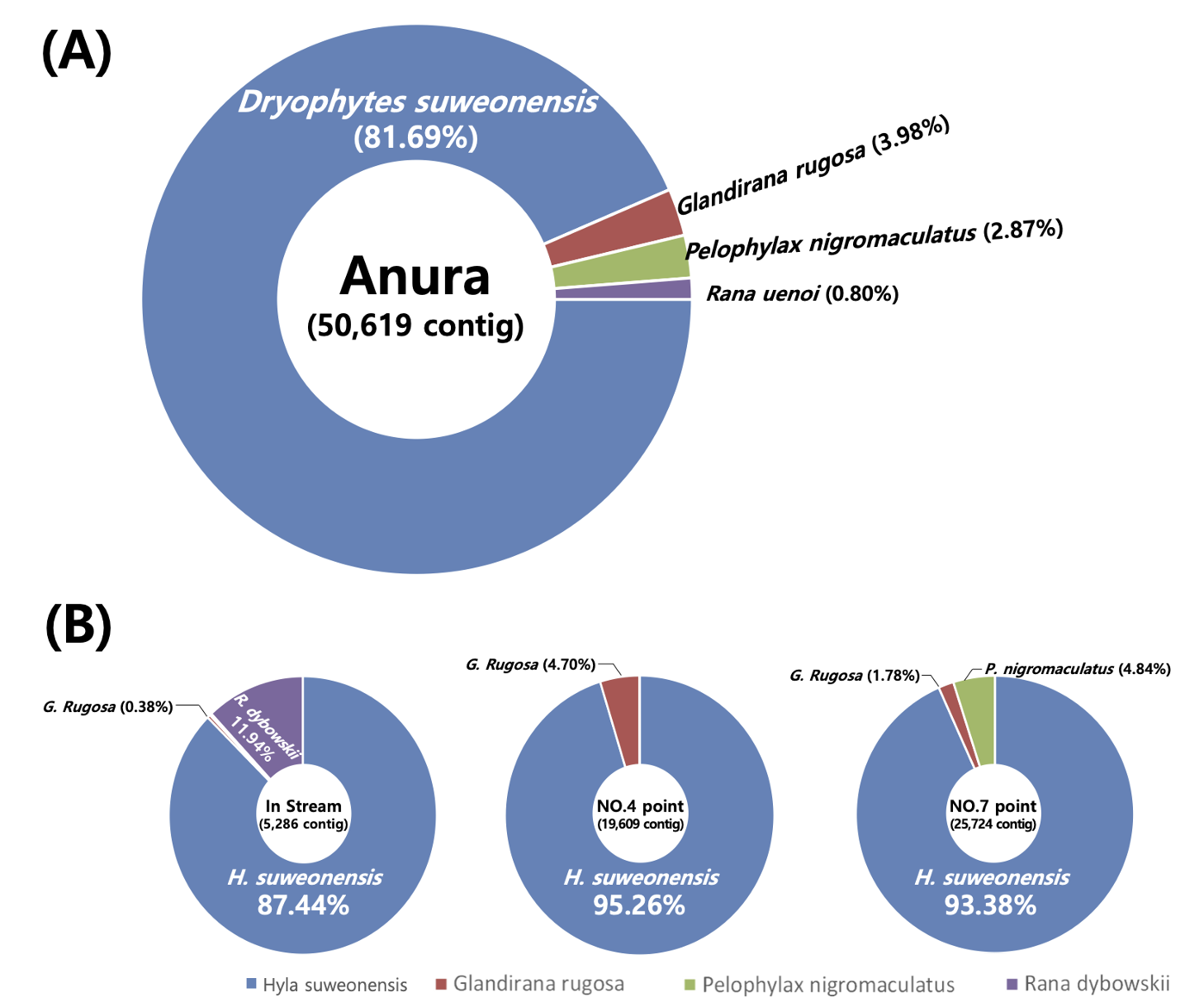
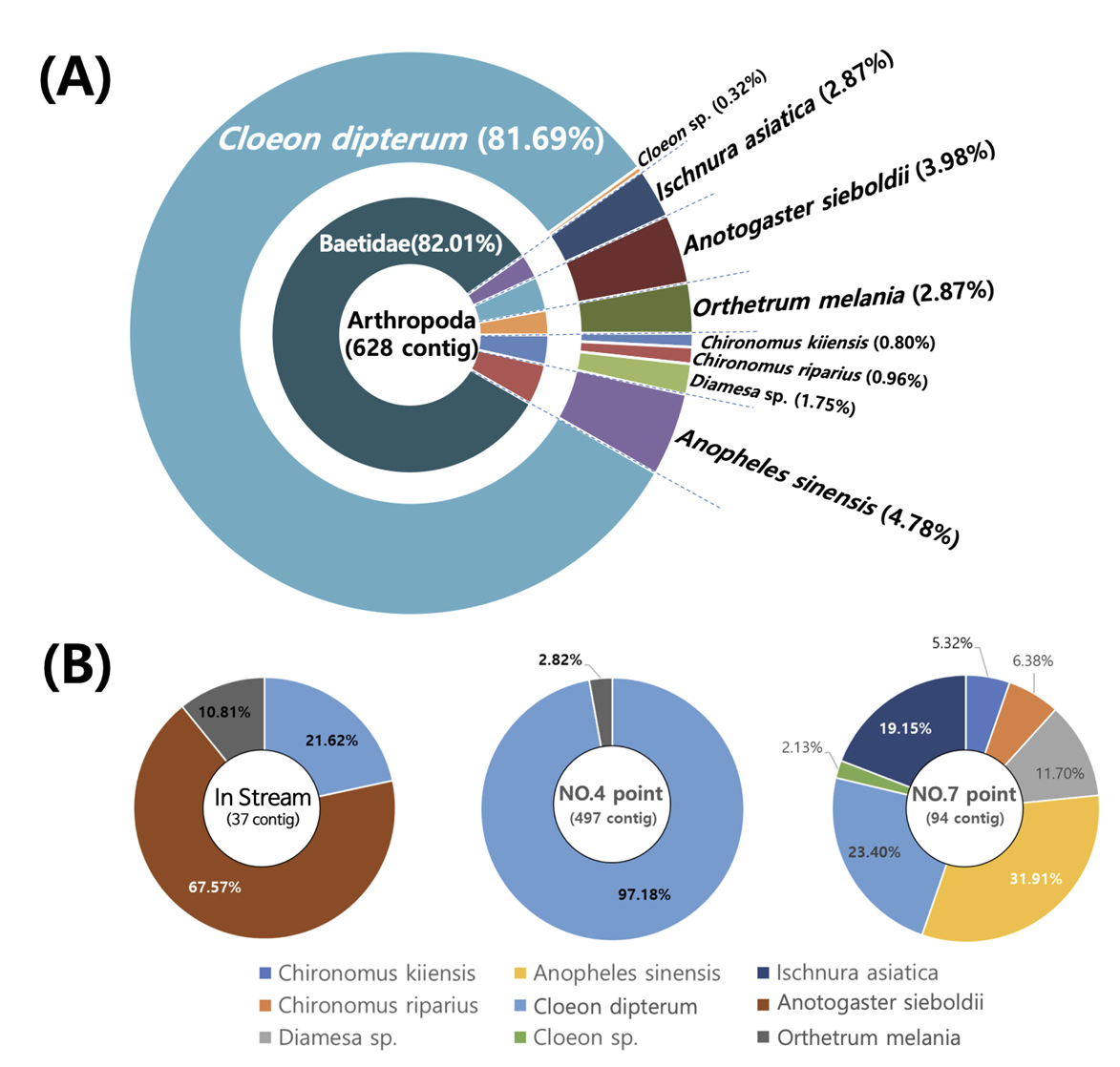
eDNA를 기반으로 논에 서식하는 개구리와 수서곤충의 유전자 염기서열을 분석한 결과, 각각 개구리는 4개 종 50,619 contig가 확인되었으며, 수서곤충은 9종 628 contig가 확인되었다(Fig. 5A).
개구리는 전체 유전자 발견지점에서 참개구리(Pelophylax nigromaculatus), 수원청개구리(Hyla suweonensis), 옴개구리(Glandirana rugosa), 큰산개구리(Rana uenoi) 가 발견되었으며, 수원청개구리의 유전자가 81.6%로 확인된 개구리 mt 16S rDNA 중에서 가장 많은 비율을 차지하였다. 참개구리, 옴개구리, 산개구리는 0.8 - 3.9% 로 상대적으로 매우 적은 비율이 발견되었다. mt 16S rDNA 유전자가 발견된 지점별로 유입수(5,286 contig)보다 논 내부 지점(평균 22,666 contig)에서 더 많은 contig가 발견되었으며 point-7에서 가장 많은 contig (25,724 contig)가 발견되었다(Fig. 5B). 개구리 유전자를 확인한 결과 수원청개구리의 유전자가 모든 point에서 80% 이상의 가장 많은 비율을 차지하였으며 옴개구리와 참개구리의 유전자가 논 내부(point-4, point-7)에서만 발견되었다. 논으로 유입되는 논수로의 유입수(pointstream) 에서는 수원청개구리 뿐만 아니라 산개구리와 옴개구리의 유전자가 발견되었으며 참개구리의 유전자는 발견되지 않았다. 이는 논 내부에서 발견된 참개구리의 유전자는 외부 유입수에서 유입된 것이 아니라 논 내부에서 기원한 것으로 판단할 수 있다.
수원청개구리는 개체의 크기가 매우 작음에도 불구하고, 상대적으로 크기가 큰 참개구리와 산개구리 보다 더 많은 비율을 차지하였다. 이는 수원청개구리의 개체수가 참개구리 및 산개구리 보다 수 배 이상 많은 경우에 발생할 수 있다(Ficetola et al., 2008; Herder et al., 2014). 다세포 생물의 특성상 하나의 개체가 살아가면서 지속적으로 세포를 환경에 유출하기 때문에 정확한 개체 수를 파악하기 어렵다(Goldberg et al., 2011; Laramie et al., 2015; Valentini et al., 2016). 하지만 이러한 개체수의 차이는 유전자의 비율 차이를 유발할 수 있기 때문에 이러한 metabarcoding 분석 결과는 주요 우점 개체를 파악하는데 중요한 정보를 제공할 수 있다. 또한 참개구리의 유전자는 point-7에서만 발견되었으며, 동일한 논 내부의 point-4 에서는 발견되지 않았다. 이는 개구리 및 어류와 같이 일정한 행동반경을 가지는 생물들의 유전자는 논 전체에서 균일하게 발생하기 보다는 일부 구간에서 주로 이루어 지며, 이로 인해 동일한 논이라 할 지라도 eDNA의 채집위치에 따라서 발견되는 종의 차이가 존재할 수 있다(Herder et al., 2013).
수서곤충은 연못하루살이(Cleon dipterum)가 81.6%로 가장 많은 비율을 차지하였으며, 또한 중국얼룩날개모기(Anopheles sinensis, 4.7%), 장수잠자리(Anotogaster sieboldii, 3.9%), 아시아실잠자리(Ischnura asiatica, 2.7%), 큰밀잠자리(Orthetrum melania, 2.8%)가 상대적으로 높은 비율을 차지하였다(Fig. 6A). 이 밖에도 연못하루살이류(Cloeon sp.), 붉은깔따구 3종(Chironomus kiiensis, Chironomus riparius, Diamesa sp.)이 확인되었다. 각 지점별로 유입수 에서는 37 contig가 발견되었으나 논 내부에서는 497 contig와 94 contig 로 논 내부에서 더 많은 contig가 발견되었다(Fig. 6B). 출현종의 유전자 염기서열을 분석한 결과, 논 내부에서 총 8개 종이 확인되었으며 논 외부의 유입수에서는 3개 종이 확인되었다(Fig. 6B). 특히 논 외부에서 발견된 장수잠자리 유전자는 논 내부에서 발견되지 않았으며, 낮은 비율을 나타낸 연못하루살이의 유전자는 논 내부의 point-4에서 97%로 가장 많은 비율을 차지하였다. Point-7은 7개 종의 유전자가 발견되었으며 중국얼룩날개모기와 연못하루살이가 50% 이상을 차지하였으며 이밖에도 아시아실잠자리와 깔따구류(Diamesa sp.)가 발견되었다. 논 내부에서 많은 비율을 차지한 중국얼룩날개모기, 아시아실잠자리, 깔따구류(Chironomus kiiensis, Chironomus riparius, Diamesa sp.) 유전자는 유입수에서 발견되지 않았다.
논 내부에서 확인된 종이 가장 적은 point-4 지점은 eDNA를 채집할 당시 수심이 낮으며, 부분적으로 논 토양이 드러나 있었다. 반면에 가장 많은 종이 확인된 point-7 지점은 수심이 상대적으로 깊고 벼로 인해 그늘이 존재하기 때문에 eDNA를 분해할 수 있는 요인들(수온변화 및 자외선)의 작용이 상대적으로 미미하였다(Barnes et al., 2014). 이러한 논 내부의 서로 다른 환경차이로 인해 하나의 논에서도 채집 위치에 따라서 종 수의 차이가 크게 나타날 수 있다. 특히 point-7 은 논 물이 배출되는 배출구와 가까운 위치로써 논 내부에 존재하는 eDNA가 물의 이동에 따라서 밀집될 가능성이 존재한다. 논에서 eDNA를 채집할 당시에는 point-7 인근의 배출구에서 논 물이 배출되고 있지 않았으나 point-7이 다른 위치에 비해 상대적으로 수량이 많고 수심이 깊은 것으로 보아, 논 물을 배출하는 시기에 point-7 지점으로 논 내부의 eDNA가 밀집된 것으로 판단된다.
Verification of eDNA detection by field survey
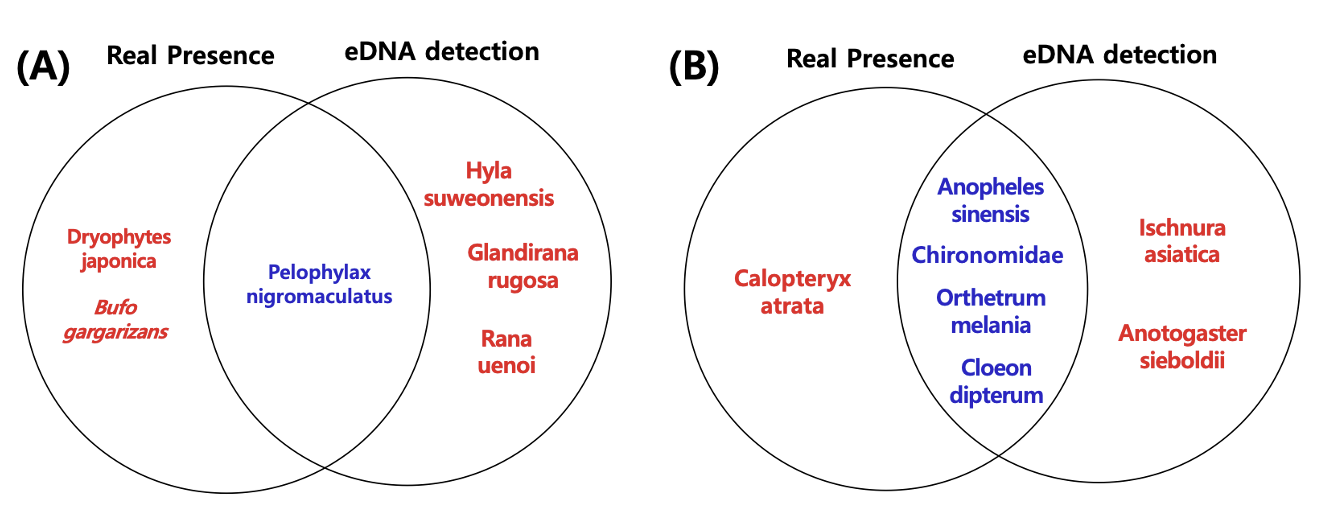
eDNA 탐색 결과 확인된 개구리 및 수서곤충의 군집을 전통적인 개체 채집방법을 통해 확인된 군집과 비교하여 eDNA를 이용한 논 생물 군집탐색 결과를 검증하였다. 전통적인 채집방법을 통해 대상 지역에서 개구리는 2종, 수서곤충은 4종을 확인하였다(Fig. 7).
대상 논 내부에서는 참개구리(P. nigromaculatus)가, 논 외부에서는 두꺼비(Bufo gargarizans) 개체가 발견되었으며, 두꺼비는 1개 개체가 발견된 반면에 참개구리는 7개체가 발견되었다(Fig. 7A). 또한 대상 지역에 거주하는 주민들을 탐문하여 개구리 사진을 확인하였으며, 이 과정에서 조사시기 이전에(5월) 청개구리(Dryophytes japonica)의 개체가 매우 많았다는 것을 확인할 수 있었다(Fig. 7A). eDNA에서 확인된 수원청개구리, 옴개구리, 산개구리는 개체가 대상 지역에서 발견되지 않았다. 대상 지역에서 eDNA와 실제 개체가 모두 발견된 종은 참개구리가 유일하였으며, 전통적인 조사방법에서는 수원청개구리, 산개구리 옴개구리 개체를 발견할 수 없었다. 두꺼비는 eDNA는 발견되지 않았으나 대상지역에서 1개 개체가 발견되었다.
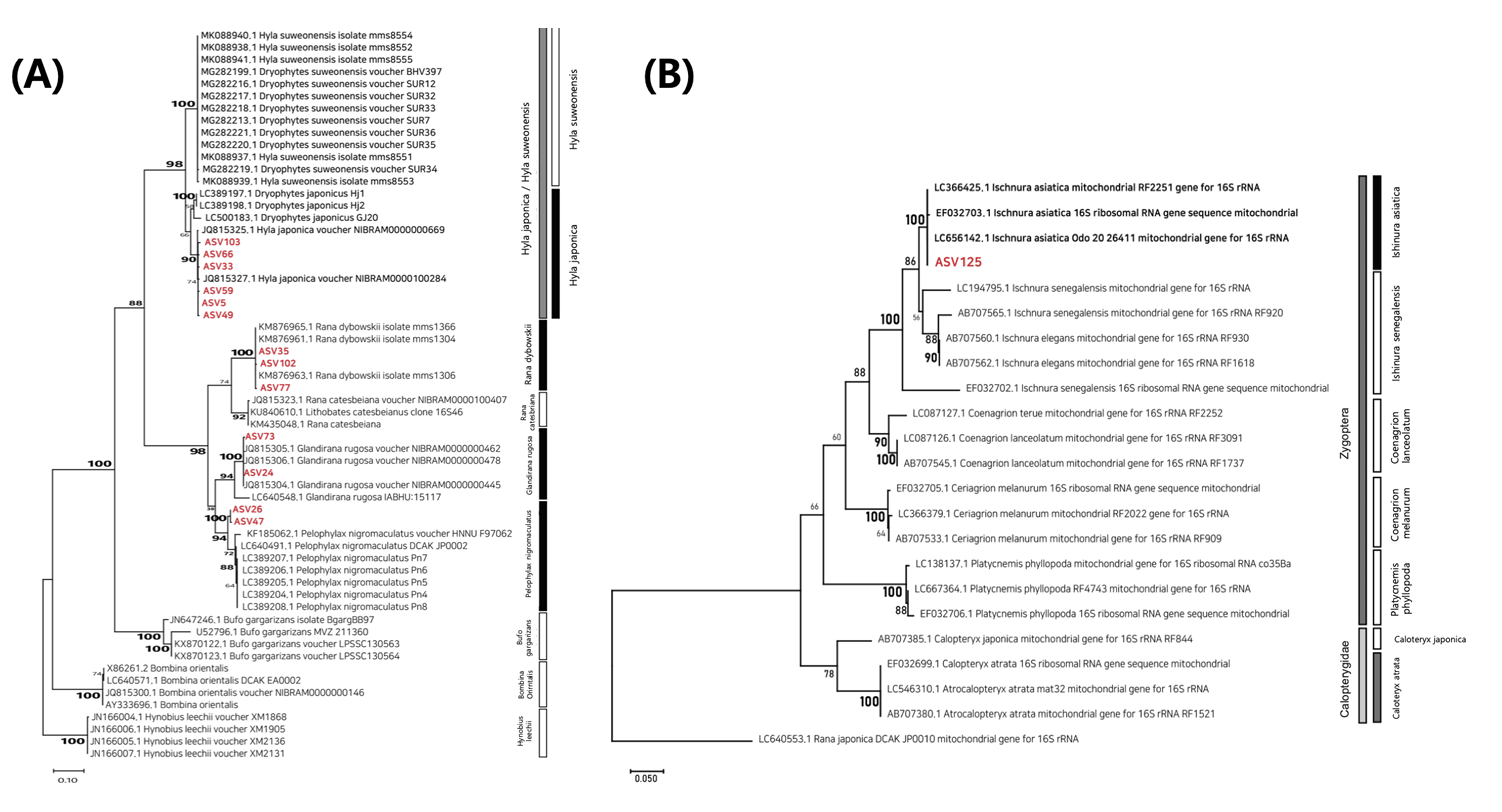
두꺼비는 주로 육상에서 기어 다니며 생활하며 산란기에만 둠벙 및 논수로에서 집단 번식한다(Günther, 1996). 산란기는 3 - 4월이며, 고인 물에 알을 낳기 때문에 현장조사가 진행된 7월에는 산란과정에서 발생한 두꺼비의 유전자가 대부분 하천으로 유출되어 논 수생태계에서 두꺼비 유전자가 발견될 가능성이 매우 적다. 또한 대상지역에서 출현한 것으로 확인된 청개구리는 성체의 크기가 약 2 - 3 cm 매우 작고, 형태적 특징이 수원청개구리와 형태적으로 매우 유사하다(Borzée et al., 2013; Kim et al., 2021). 청개구리와 수원청개구리를 구분하는 특징은 물갈퀴의 차이, 울음주머니의 색, 울음소리 등으로 전문적인 동정 지식이 없다면 수원청개구리와 청개구리를 구분하는 것은 매우 어렵다(Ham, 2014). 또한 수원청개구리는 평야지대의 논과 주변의 농수로에서 서식하기 때문에 본 연구지역과 같이 산으로 둘러 쌓인 소규모 논은 수원청개구리가 서식하기 적합하지 않은 지역이다(Borzée and Jang, 2015). 본 연구에서 발견된 수원청개구리의 DNA 염기서열은 계통 분석하였을 때 수원청개구리 보다는 청개구리의 유전자와 단계통을 이루었으며, Hyla japonicus 와 90%의 확률로 하나의 단계통을 이루었다(Fig. 8A). 이와 같이 염기서열의 유사성으로 인해 두 분류군이 오동정(miss identification)되는 경우가 존재할 수 있으며 이러한 이유 때문에 유전자 분석결과의 실제 검증 필요성이 더욱 높은 것으로 판단된다. 또한 대상 분류군의 성체가 이미 다른 지역으로 이동하거나, 산란 후 개체를 찾아볼 수 없는 수준이더라도 논 환경에 존재하는 eDNA에는 대상 분류군의 유전물질이 존재하게 된다. 따라서 eDNA를 이용한 생물탐색은 대상 지역에 서식하고 있는 생물뿐만 아니라 대상 지역을 거쳐간 생물들까지 포함하여 군집을 파악할 수 있다.
대상 논 내부와 인근에서 수서곤충은 연못하루살이(Cloeon dipterum), 깔따구류(Chironomidae) 및 중국얼룩날개모기(Anopheles sinensis), 검은물잠자리(Calopteryx atrata)가 발견되었다(Fig. 7B). 연못하루살이는 논 내부에서 유충 상태의 8개체를 발견하였으며, 깔따구류와 중국얼룩날개모기는 성충이 되어 비행하는 개체를 다수 확인하였다(Fig. 7B). 3개 분류군 모두 eDNA기반의 군집분석에서 유전자를 확인할 수 있었다. 반면에 검은물잠자리는 1개체가 발견되었으며 eDNA 탐색과정에서 유전자가 발견되지 않았다. eDNA 탐색과정에서 유전자가 발견된 아시아실잠자리(Ischnura asiatica)와 장수잠자리(Anotogaster sieboldii)는 대상 논에서 유충과 성충 모두 발견되지 않았다. 큰밀잠자리(Orthetrum melania)는 조사기간에는 유충과 성충 모두 발견되지 않았으나 조사이후 대상 지역에서 성충의 비행을 확인할 수 있었다. 결과적으로 깔따구류, 중국얼룩날개모기, 연못하루살이, 큰밀잠자리만 대상 지역에서 eDNA 와 개체가 모두 확인되었으며 수중에 서식하는 유충은 연못하루살이만 확인되었다.
깔따구류 및 중국얼룩날개모기의 유충은 이동성이 없으며, 물 표면에 부유하여 생활한다(Hinne et al., 2021; Karima, 2021). 따라서 전통적인 트랩 채집법으로 이동성이 없는 깔따구류와 중국얼룩날개모기의 유충을 채집하는 것은 매우 어렵다. 반면에 eDNA는 유충이 서식하면서 발생하는 유전물질을 탐색하기 때문에 향후 둠벙 및 논두렁의 모기유충을 매우 효율적으로 탐색할 수 있다.
eDNA 탐색과정에서 발견되지 않은 검은물잠자리는 물잠자리과(Calopterygoidea)에 속하는 분류군으로써 실잠자리과(Coenagrionidae) 분류군과 함께 실잠자리아목(Zygoptera)에 포함된다. 본 연구에서 증폭된 mt 16S rDNA 염기서열 구간은 실잠자리과와 물잠자리과를 구분할 수 있는 특이성이 존재하며 대상 논에서 발견된 아시아실잠자리의 mt 16S rDNA 유전자가 검은물잠자리의 유전자일 가능성은 매우 낮다(Fig. 8B). 일반적으로 큰밀잠자리의 비행은 주로 6 - 8월 사이에 발생하며, 특히 7월에 집중된다(Kim, 2015; Lee, 2019). 하지만 본 연구에서 큰밀잠자리 성충의 비행은 주로 7월 말 - 8월에 발견되었으며 비행하는 성충의 수가 매우 적었다. 이는 2022년 6 - 7월 중에 발생한 집중 강우로 인해 잠자리 유충이 물 밖으로 나오기 어려운 환경이 조성되었으며 이로 인해 유충의 우화시기가 늦어진 것으로 판단된다. 이러한 집중 강우는 논에 서식하는 생물들의 생물계절(phenology)에 영향을 미칠 수 있으며(Barrett and Brown, 2021; Groffen et al., 2022; Henry et al., 2022; Numata et al., 2022), 이는 전통적인 조사방법으로는 개체를 확인하기 어려울 수 있다. 하지만 대상 생물이 서식하였던 환경에서는 대상 생물의 유전자가 분포하며, eDNA를 이용한 생물 탐색은 이러한 논 생물의 서식 흔적을 탐색하기 때문에 개체가 직접적으로 발견되지 않았으나 eDNA 기반의 생물탐색을 통한 유전자 수준의 흔적 탐색이 가능하다.
Fig. 8
Phylogenetic tree of (A) Anura and (B) Zygoptera mitochondrial 16S rDNA. The evolutionary history was inferred by using the Maximum Likelihood method and Tamura-Nei model. The percentage of trees in which the associated taxa clustered together is shown next to the branches. This analysis involved 60 nucleotide sequences. There were a total of 271 positions in the final dataset. Evolutionary analyses were conducted in MEGA11. Red color text mean amplicon in this study.
Conclusion
논 수생태계는 하천 및 저수지와 연결되어 있어서 개구리 및 수서곤충 등의 산란과 서식장소로 이용되고 있다. 따라서 논 환경의 eDNA에 존재하는 생물들의 유전자 염기서열을 분석하여 논 생태계에 서식하는 다양한 생물들의 존재를 파악할 수 있다. 본 연구에서도 현장 조사과정에서 발견되지 않은 수원청개구리 및 큰밀잠자리의 유전자를 탐색함으로써 성체 생활시기가 다르거나 성충으로 우화하여 대상 생태계에서 더 이상 발견할 수 없는 분류군까지 존재 여부를 간접적으로 파악할 수 있었다. 특히 대상 지역 뿐만 아니라 논과 인접하여 생활하는 생물들 까지도 존재 여부를 파악할 수 있으며 논과 연결되어 있는 상류 및 논수로에서 서식하는 생물들의 존재도 파악할 수 있다. 이러한 eDNA 기반의 논 생물탐색은 강력한 분석 해상도(resolution) 때문에 농업 생태계 생물다양성 조사에서 활용가치가 매우 높을 것으로 판단된다. 하지만 eDNA 탐색 결과는 대상 지역에 서식하는 생물들의 흔적을 분석하는 기법으로써 논 생태계 특성상 매우 다양한 외부 오염이 발생하기 용이하며, 이로 인해 외부에서 유입된 유전자가 대상 지역의 생물 군집 분석에 영향을 미칠 수 있다. 따라서 eDNA 기반의 생물 군집 조사와 실제 개체조사는 서로 별개의 영역이 아닌, 서로 상호작용을 할 수 있는 하나의 영역으로 구분해야 한다. 즉, eDNA 탐색 결과를 기반으로 대상 지역에서 출현예상 종목록을 도출하고, 이를 전통적인 개체조사를 통해 검증함으로써 형태적인 분석에서 확인하기 어려운 교잡종의 출현 및 돌연변이 발생을 파악할 수 있다. 또한 eDNA 탐색에서 발견되지 않은 개체가 전통적인 개체조사에서 발견되었을 때 이에 대한 원인을 분석하고 이를 통해 eDNA 채집방법 및 유전자 증폭구간을 개선할 수 있다. 결과적으로 이러한 eDNA와 전통적인 조사방법의 상호 보완적인 관계는 두 조사방법의 저변을 확대하는데 매우 긍정적인 영향을 미칠 것으로 판단된다.
Summary
논(paddy) 환경은 습지로 분류되며 담수환경에서 매우 많은 비율을 차지하고 있다. 또한 많은 수서곤충류 및 양서파충류 유생의 서식지 및 산란 장소로써 생태학적으로 매우 중요하다. 하지만 기후변화 및 무분별한 농약 살포 등으로 인해 논 생태계는 지속적으로 위협받고 있다. 따라서 향후 훼손된 논 생태계를 복원하기 위해서는 복원 기준이 될 수 있는 논 생태계 서식 생물들의 정보가 필요하다. 환경유전자 metabarcoding 분석법은 성체 생활시기가 다르거나 성충으로 우화하여 대상 생태계에서 더 이상 발견할 수 없는 분류군까지 존재 여부를 간접적으로 파악할 수 있기 때문에 논 생태계에 서식하는 많은 생물들의 정보를 축적하는데 매우 효과적이다. 본 연구에서도 4종의 개구리와 9종의 수서곤충 유전자가 발견되었으며, 일부 분류군은 현장에서 개체가 직접 발견되었다. 많은 수의 분류군이 DNA 탐색에서만 발견되었으며, 전통적인 조사방법은 매우 제한적인 분류군만을 확인할 수 있었다. 이러한 eDNA 기반의 논 생물탐색은 강력한 분석 해상도 때문에 농업 생태계 생물다양성 조사에서 활용가치가 매우 높을 것으로 판단된다.